20230210 如果Pol II想要跑,稳定G4可以留

#
引言
转录因子、染色质相关蛋白、组蛋白修饰和DNA甲基化等作为表观遗传学明星已被研究许多年,除此之外,DNA二级结构也存在转录调控功能,其中研究最多的便是G4结构。G4结构主要在基因启动子区形成并提升基因表达水平。本次带来的文献与康测科技新开发的G4-ChIP-seq产品有关,该文献从染色质状态入手,主要讨论启动子区G4结构在不同染色质状态下的变化情况以及RNA聚合酶II(Pol II)的结合情况。
#
背景
G4结构在人基因组中主要定位于启动子区,G4-ChIP数据显示内源性G4结构富集在转录起始位点(TSS)上游的无核小体区(nucleosome-depleted region, NDR)。如果某个基因启动子区存在内源G4结构会具有更高的转录活性。组蛋白去乙酰化酶抑制剂导致的染色质松弛(Chromatin relaxation)也会增加G4结构的形成。由于超过99%的内源性G4结构与转录因子(TF)结合位点重叠,这种基因表达水平升高可能是由于启动子区G4结构的TF占据增加和招募Pol II引起的。基于前文的结果,那么具有转录活性的基因启动子处的G4结构形成是转录活性增加的原因还是结果?作者因此开展G4-ChIP-seq、RNA Pol II ChIP-seq和ATAC-seq来回答这个问题。
#
研究内容
1. G4结构形成是Pol II结合启动子增加的标志
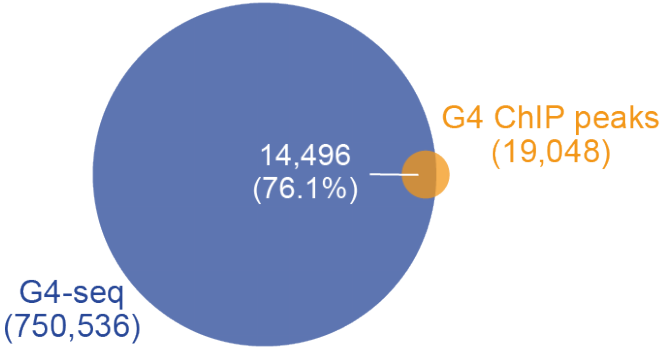
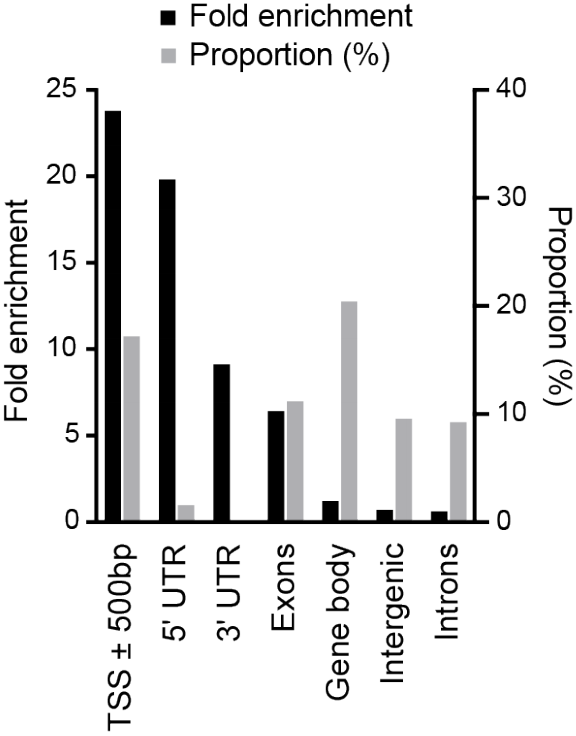
作者首先使用K562细胞作为研究对象,使用G4-ChIP-seq、Pol II ChIP-seq和ATAC-seq分别鉴定G4结构位置、Pol II位置以及染色质开放区域。G4-ChIP鉴定到的peak可以使用DNA聚合酶停滞试验(G4-seq)来验证,结果显示76.1%的peak可以在体外形成G4结构(下图左);作者定义TSS上下游500 bp为启动子区,而G4 peak主要在启动子区富集(下图右)。与数据库中转录组数据联合分析发现,启动子区具有G4结构标记的基因表达水平显着增加。
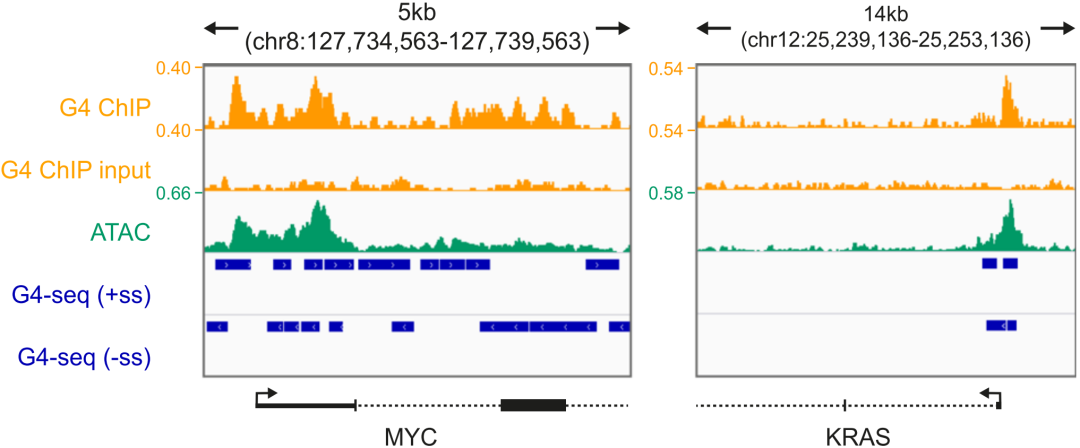
ATAC-seq结果显示G4结构(88.2%)大部分处于NDR区域(染色质可及区),例如MYC和KRAS启动子区(下图)。
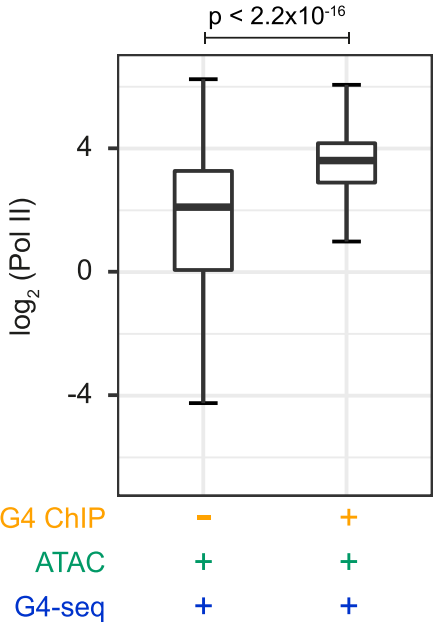
Pol II ChIP-seq结果显示相比无G4结构的启动子,G4启动子区具有较多的Pol II占位(下图)。
为了阐明G4结构形成是否受到活跃的转录状态的影响,以及G4结构形成是否受更开放与更紧凑的染色质环境调控,作者将集中研究Pol II占位的G4启动子。
2. 启动子区G4结构形成不依赖活跃的转录状态
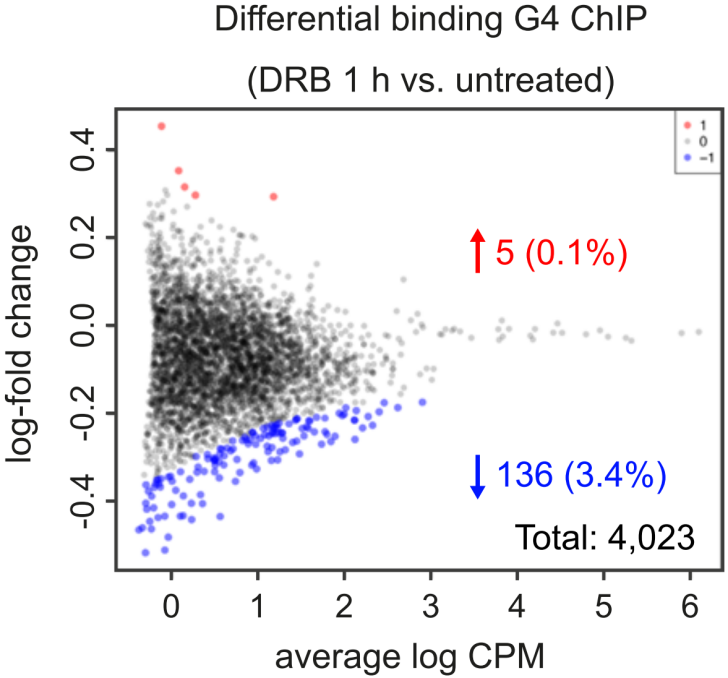
为了研究G4结构形成是否依赖活跃的转录状态,作者使用DRB抑制转录延伸过程,来看转录抑制是否可以造成启动子区G4结构的消失。将DRB处理和DMSO处理的K562细胞同时进行G4-ChIP-seq,结果显示启动子区G4结构在DRB处理后并没有发生显著变化(下图)。因此抑制Pol II依赖性延伸过程不会导致启动子处G4结构的消失,说明G4结构形成是在转录延伸之前。
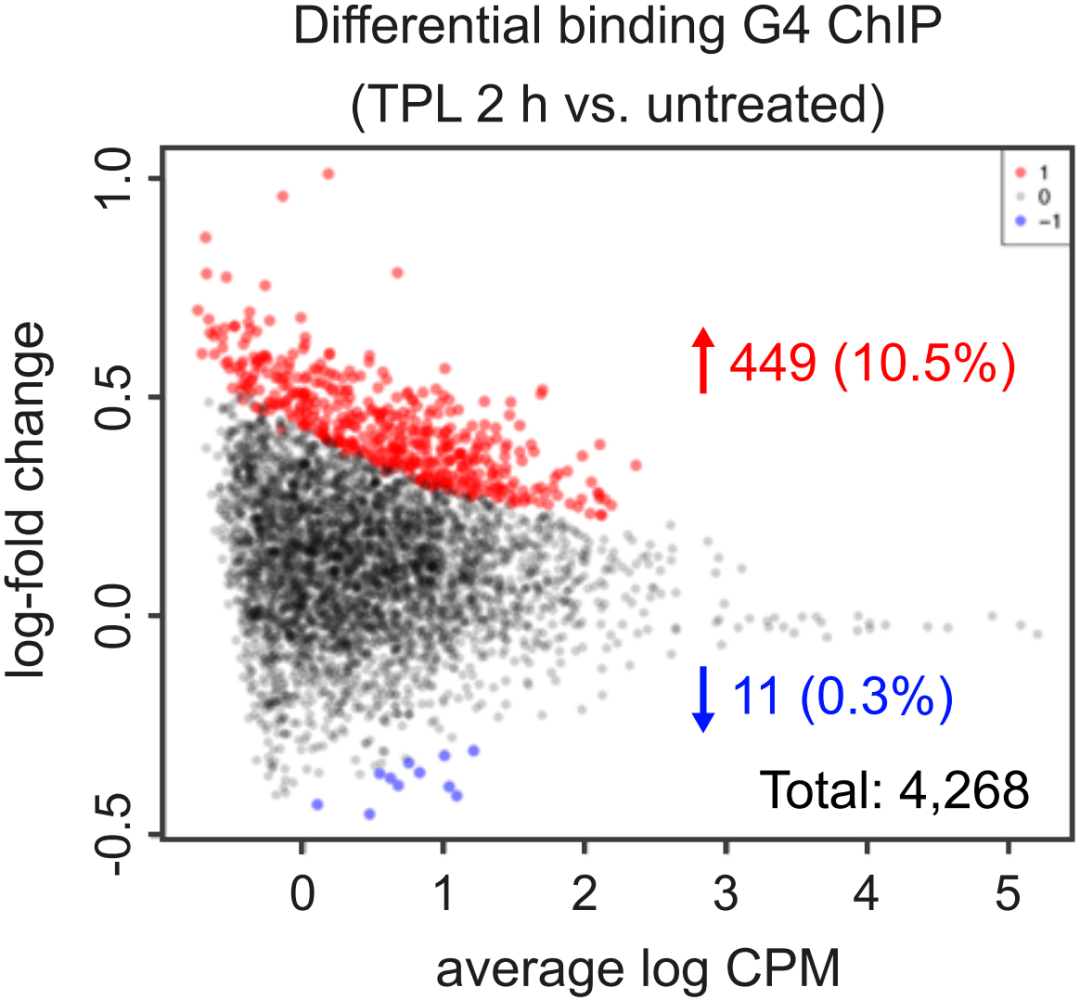
为了评估抑制转录起始过程是否会导致启动子G4结构消失,作者使用TPL(共价抑制Pol II相关的解旋酶XPB)处理后进行G4-ChIP-seq,结果显示TPL抑制转录起始过程并未显着降低整体启动子G4的信号;反而TPL处理后会使启动子区G4信号增加(下图)。作者的结果与早期研究结果相反(活跃转录状态可以产生更多的G4结构),作者证明活跃转录状态与启动子区G4结构形成无关。
3. 启动子区G4结构折叠对染色体压缩敏感
考虑到转录抑制不能去除启动子区的G4结构,而且抑制组蛋白去乙酰化酶活性造成染色质松弛可以增加G4结构的形成,作者假设染色质开放状态会影响G4结构的形成。使用缺氧诱导染色质压缩(H3K9me3修饰水平上升,组蛋白乙酰化水平下降),对缺氧处理和常氧对照的K562细胞进行ATAC-seq和G4-ChIP-seq,结果显示常氧状态下,相比无G4结构的启动子(Pol II+ G4+),具有G4结构的启动子(Pol II+ G4+)定位在跟开发的染色质区域(下图)。

诱导缺氧状态下可以导致大多数G4启动子区域的ATAC-seq信号强度显着降低(下图)。

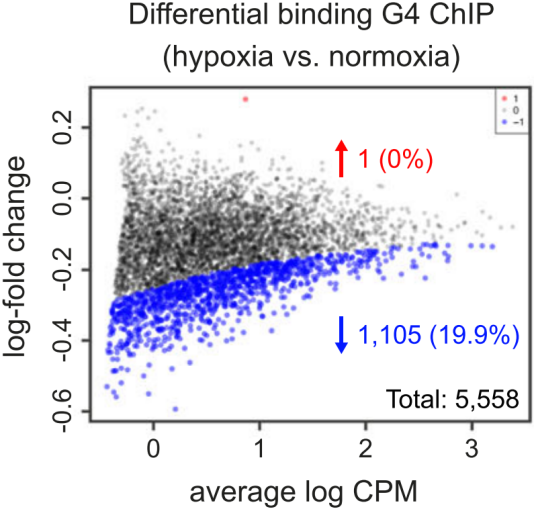
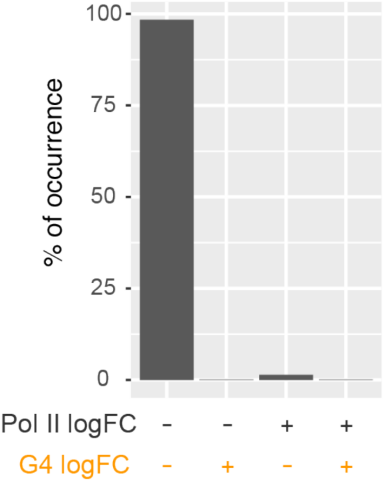
诱导缺氧状态下,20%左右的启动子区G4的信号显着降低(下图),这说明许多启动子区G4结构对染色质压缩敏感。
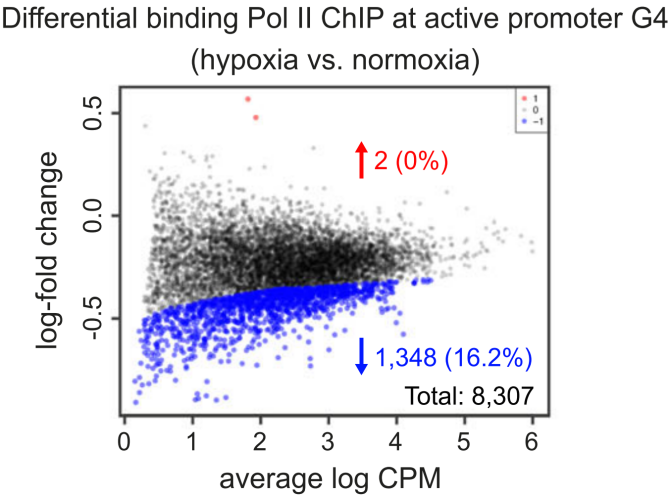
另外,缺氧状态下的具有G4启动子区的Poll II信号强度整体下降(下图左),Pol II信号丢失几乎完全发生在G4结构减少的区域(下图右)。
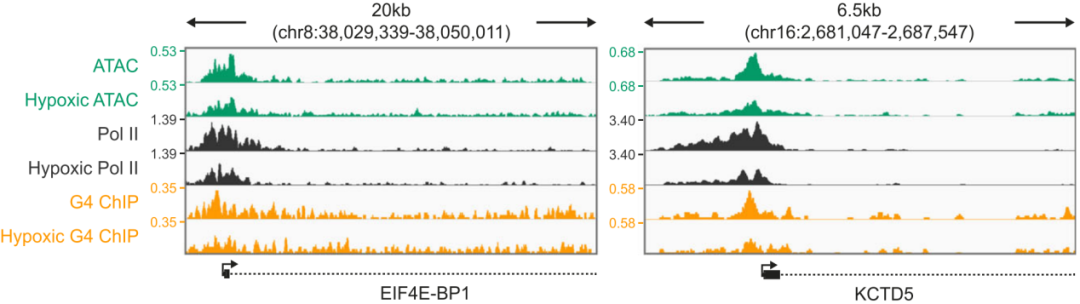
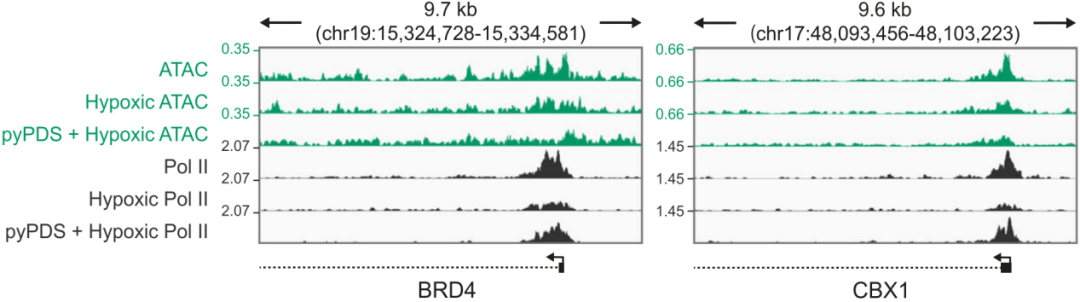
具体基因的各组学reads覆盖度图也能佐证(下图),说明染色质压缩导致许多启动子区G4结构的损失,并伴随Pol II结合的丢失。
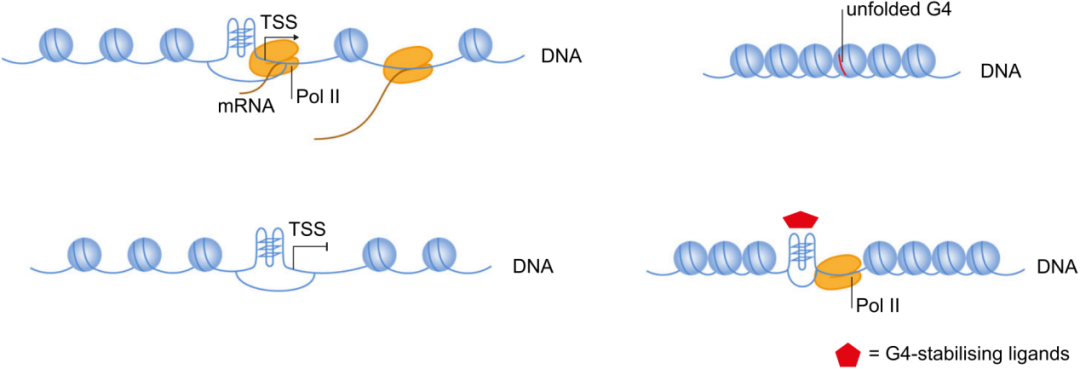
4. 通过小分子稳定G4结构可以抵消缺氧状态下G4结构和Pol II信号丢失
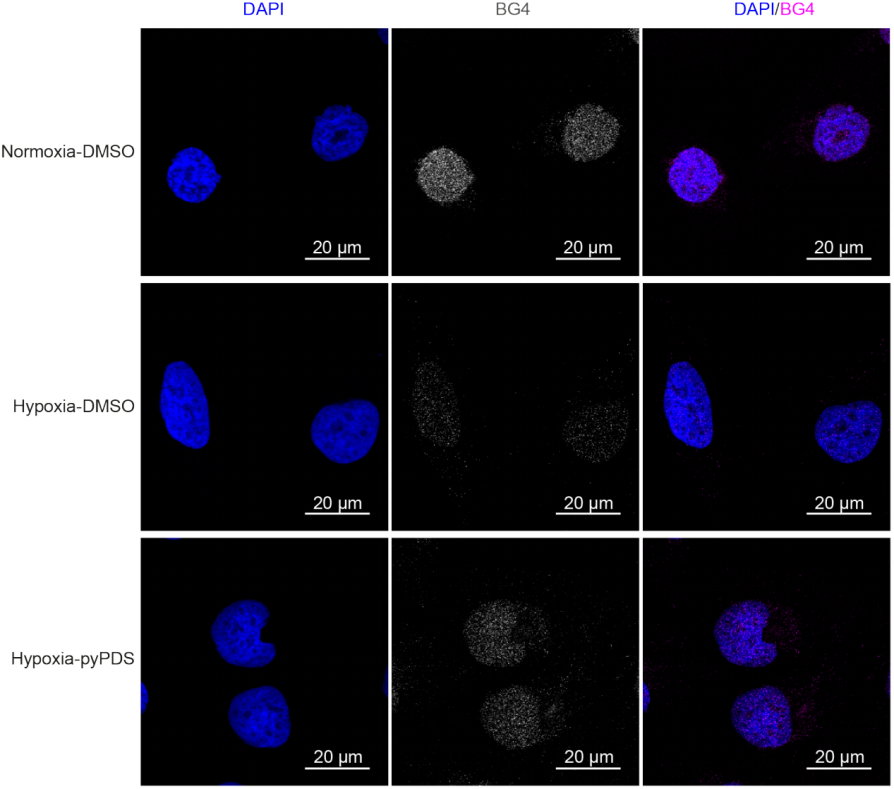
为了确定G4结构稳定性是否影响缺氧状态下G4的形成,作者使用小分子pyPDS来稳定G4结构,核成像结果显示G4信号明显增加,说明pyPDS可以阻止低氧状态下G4结构的解开。
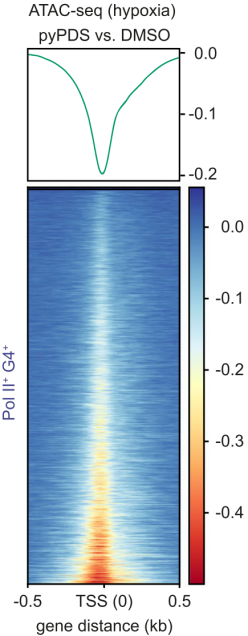
作者首先利用ATAC-seq确定pyPDS处理不会明显改变缺氧状态下的染色质可及性,结果显示染色质大部分还是处于压缩状态下,如下图。
下一步作者观察缺氧条件下,稳定的G4结构能否留住Pol II的结合,ChIP结果显示pyPDS处理可以使缺氧条件下Pol II结合损失减少10倍(下图上),例如BRD4和CBX1(下图下)。
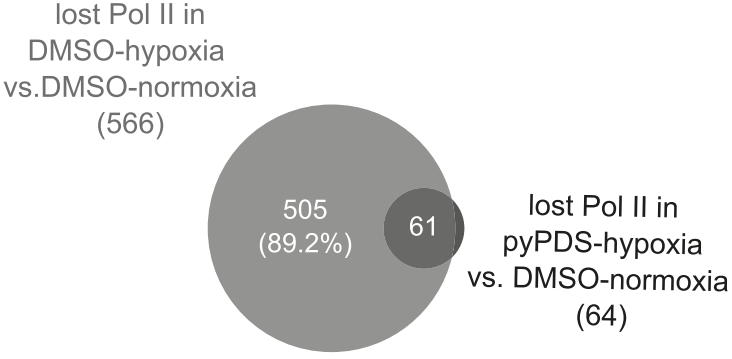
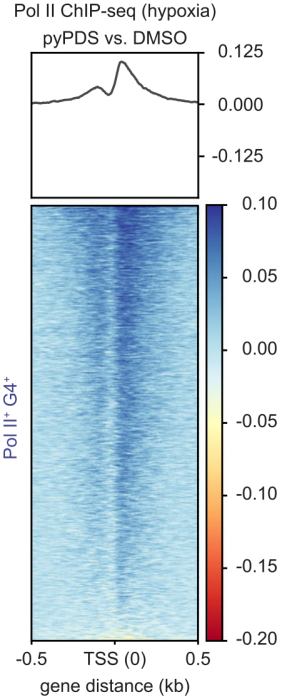
另外在常氧状态下pyPDS处理或缺氧状态下非G4结构启动子区Poll II结合没有发生明显改变(下图)。
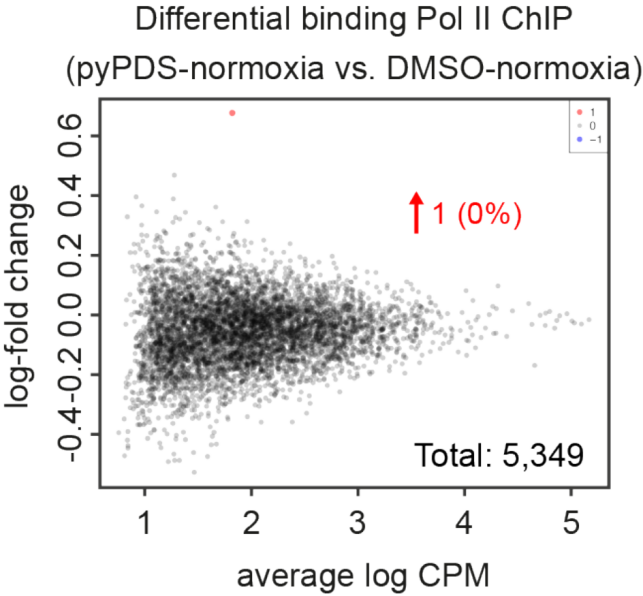
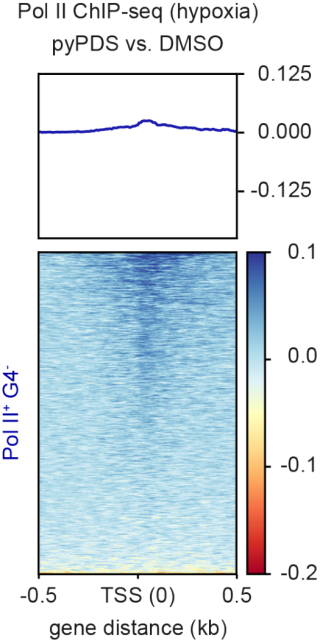
这些结果共同说明在染色质压缩过程中维持G4结构可以保留Pol II的结合。
#
总结
作者利用G4-ChIP-seq、Pol II ChIP-seq和ATAC-seq,从染色质状态入手,探讨启动子区G4结构的形成和变化情况,结果说明G4结构形成在转录起始之前,并且染色质压缩会导致G4结构的消失,让我们总结下作者的工作:
1. 通过G4-ChIP-seq、Pol II ChIP-seq和ATAC-seq确定K562细胞G4结构定位、Pol II结合情况以及染色质可及性状态;
2. 分别使用DRB(抑制转录延伸)和TPL(抑制转录起始)处理后,通过G4-ChIP-seq发现G4结构形成不受活跃转录状态影响;
3. 利用缺氧处理K562细胞造成染色质状态全局压缩,通过ATAC-seq确认染色质可及性降低,再通过G4-ChIP-seq发现启动子区G4结构大部分消失;同时Pol II ChIP-seq结果显示G4结构启动子区Pol II结合变少;
4. 最后通过小分子G4结构稳定剂pyPDS处理,在缺氧状态下维持启动子区G4结构可以保留Pol II在启动子中的结合。
#
参考文献
Shen, J., Varshney, D., Simeone, A. et al. Promoter G-quadruplex folding precedes transcription and is controlled by chromatin. Genome Biol 22, 143 (2021).
DOI: 10.1186/s13059-021-02346-7











