-
-
-
核心技术 >
检测服务 >
产品/解决方案>
-
RNA上目前发现存在着170多种不同类型的转录后修饰,并且所有已知的RNA种类都可以被修饰,其中rRNA和tRNA的修饰最多。mRNA上的主要修饰类型包括m6A、m1A和m5C。m5C修饰是最早发现的RNA修饰之一,最初研究表明主要分布在tRNA和rRNA上,近期研究表明,真核生物的mRNA上也存在m5C修饰,其可以维持mRNA的结构和稳定性、调控翻译过程等。与m6A修饰类似,对m5C进行研究同样绕不开催化/擦除m5C修饰的修饰酶(Writers)/去修饰酶(Erasers),以及各种阅读器蛋白(Readers),已经有研究证明具有m5C修饰的mRNA的命运可以由m5C修饰的reader蛋白调节,而且在某些病理情况下,可以使mRNA的m5C修饰水平错误调节。
Writers:
·NSUN2是大部分mRNA和非编码RNA上的m5C修饰的催化酶;
·NSUN家族其余成员行使各自的催化酶功能,具体如下表;
·DNMT2主要负责真核生物天冬氨酸(Asp)tRNA m5C修饰的催化酶,修饰主要发生在反密码子环中的C38处;
·TRM4A/TRM4B是在植物拟南芥中鉴定出的催化酶。
Erasers:
·TET1/TET2/TET3主要负责mRNA去甲基化酶;
·ALKBH1功能见下表;
Readers:
·ALYREF和YBX1均负责识别mRNA上的m5C修饰。

m5C meRIP-seq
meRIP-seq是参考Chip-seq发展出来的鉴定RNA修饰的一种方法,可以广泛应用于RNA修饰的研究,只要目标修饰存在商业化抗体,就可以针对该修饰进行meRIP-seq研究。目前meRIP-seq已成功应用于:
m1A修饰(m1A-seq)
m5C修饰(m5C-seq)
meRIP-seq的实验原理如下图所示:首先通过polyA捕获mRNA,或者通过rRNA剔除去掉rRNA,然后将获得的RNA打成100nt左右的小片段;之后使用特定修饰特异的抗体(以m5C抗体为例)进行免疫共沉淀,含有m5C修饰的RNA片段被富集并回收;进而对回收的RNA进行文库构建、高通量测序和生物信息学分析,通过peak分析鉴定出m5C修饰的位点。
由于rRNA中含有m5C修饰,因此实验过程中rRNA的剔除是必需的。目前存在两类技术去除rRNA的干扰,一种是polyA捕获正筛mRNA、另一种是rRNA剔除负筛rRNA,这两种方法成本上存在着巨大的差异,同时其检测对象和结果也显著不同:
mRNA捕获:便宜,但仅能对mRNA和部分带有polyA尾的lncRNA上的m5C修饰进行研究,是目前最常用的方法;
rRNA剔除:贵,且受物种限制(参考这里),但该方法可以全面检测mRNA、lncRNA、circRNA和pre-mRNA上的m5C修饰,是非常全面的方法;

nano m5C meRIP-seq
与nano m6A meRIP类似,nano m5C meRIP技术可以解决一些特殊样本如少量原代细胞、珍贵的临床样本等无法获得足量的RNA的问题,实验流程如下:
- 首先将Total RNA进行片段化,至100 nt长度;
- 使用m5C抗体进行IP,并回收RNA;
- 将IP下来的RNA全部用于文库构建:由于rRNA上带有m5C修饰,因此该RNA中含有大量的rRNA,大幅提高了RNA量,使文库构建得以成功;
- 将构建好的文库,使用DSN消化高丰度的rRNA来源的DNA,从而富集mRNA、lncRNA和circRNA;
- 高通量测序和生物信息学分析
本技术可以针对低至500 ng的Total RNA进行meRIP-seq研究,同时还可以检测lncRNA、circRNA甚至是病毒RNA的的m5C修饰情况。
服务流程
您只需要提供足量的组织/细胞或RNA,我们将根据物种、RNA起始量为您推荐合适的meRIP实验方案~

产品优势
为什么选择我们?
数百个项目经验!
UMI RNA-seq建库加成!

实测数据
Peak在mRNA上的分布
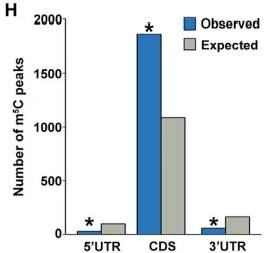
目前mRNA的m5C修饰研究还相对较少,人、小鼠等m5C修饰分布在mRNA的5’UTR、CDS和3’UTR区域,而且在start codon处有特异性peak;拟南芥中主要分布在CDS和3’UTR区域,而且在stop codon处有特异性peak,因此m5C修饰的分布具有物种特异性。

Peak Reads在基因上的分布

文章解析
拟南芥中的RNA m5C甲基化修饰
m5C是一种研究较为深入的DNA修饰方式,并且主要报道见于原核与真核的ncRNA中。然而,在植物mRNA中m5C的分布与功能仍并不被人所知。在这项研究中通过m5C-RIP-seq技术研究了拟南芥中m5C RNA的全基因组特征。
首先通过LC-MS/MS和Dot blot分析对拟南芥m5C的整体特征进行鉴定,m5C修饰在各种RNA中均存在,且在mRNA的修饰占高比例;m5C修饰在各组织中也普遍存在,且在角果中占高比例;随着发育的进行,m5C修饰比例逐渐增加(下图C, E)。

随后通过m5C-RIP-seq对拟南芥幼苗进行全转录组的m5C分析,得到6045个m5C peaks,并通过m5C-RIP-qPCR进行验证测序结果(下图D)。RNA-seq数据和m5C-RIP-seq数据联合分析表明,无论是高表达还是低表达的转录本均能发现m5C的修饰(下图C)。

通过体外的实验证实TRM4B是RNA m5C修饰的甲基转移酶。取tum4b突变株进行m5C-RIP-seq,m5C的特征与前述正常拟南芥测序相一致,但是m5C修饰明显减少(下图A-H)。

参考文献:
Cui X, Liang Z, Shen L, et al. 5-Methylcytosine RNA Methylation in Arabidopsis Thaliana.[J].Molecular Plant, 2017, 10:1387.
m5C meRIP-seq
联系我们
地址:武汉市江夏区东湖新技术开发区高新六路99号南山·光谷自贸港A栋6楼
邮箱:seqhealth@seqhealth.cn
电话:027-65527552
热门服务
关注康测公众号











