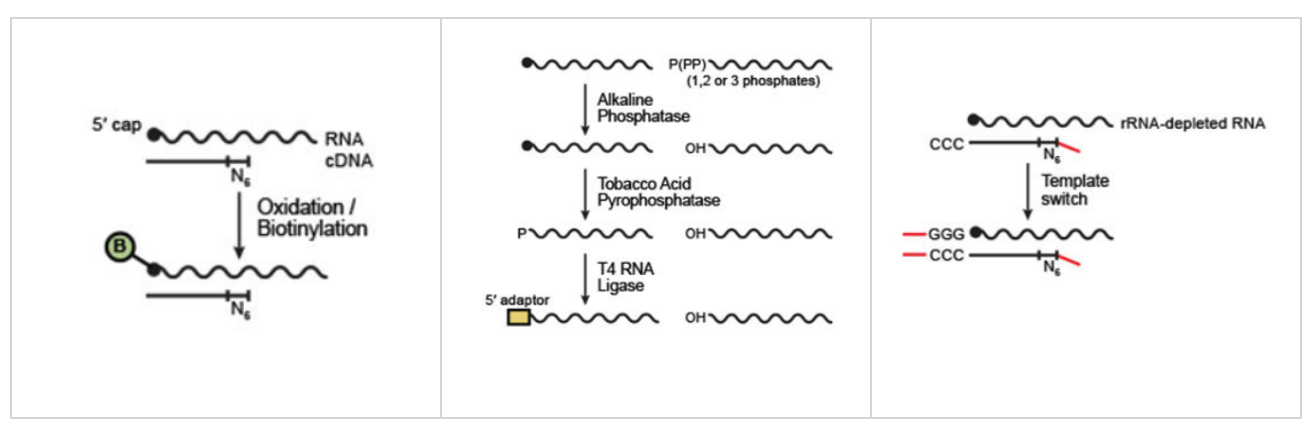
真核生物mRNA的成熟需要经历三个主要的加工过程:剪接、5'加帽、3‘加尾;而5‘加帽则是其中第一个加工步骤:在RNA被转录出25-30个碱基就会开始加帽修饰。因此,成熟mRNA的5’末端都带有m7G帽子结构。
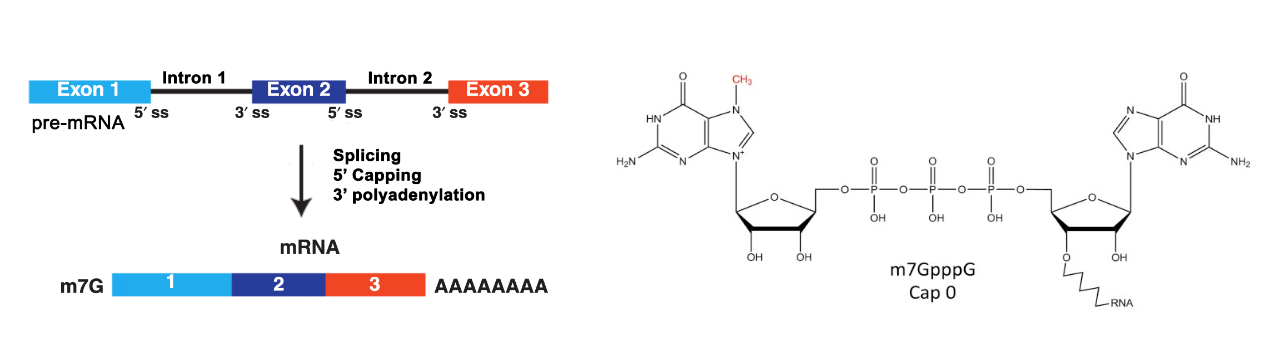
mRNA的5‘ cap具有重要的生物学功能:它一方面可以保护RNA免于5’-3‘核酸外切酶降解,另一方面作为一个锚点为剪接、加尾以及出核募集相关蛋白。而5‘ cap最重要的生物学功能,则是其在翻译起始过程中的作用。
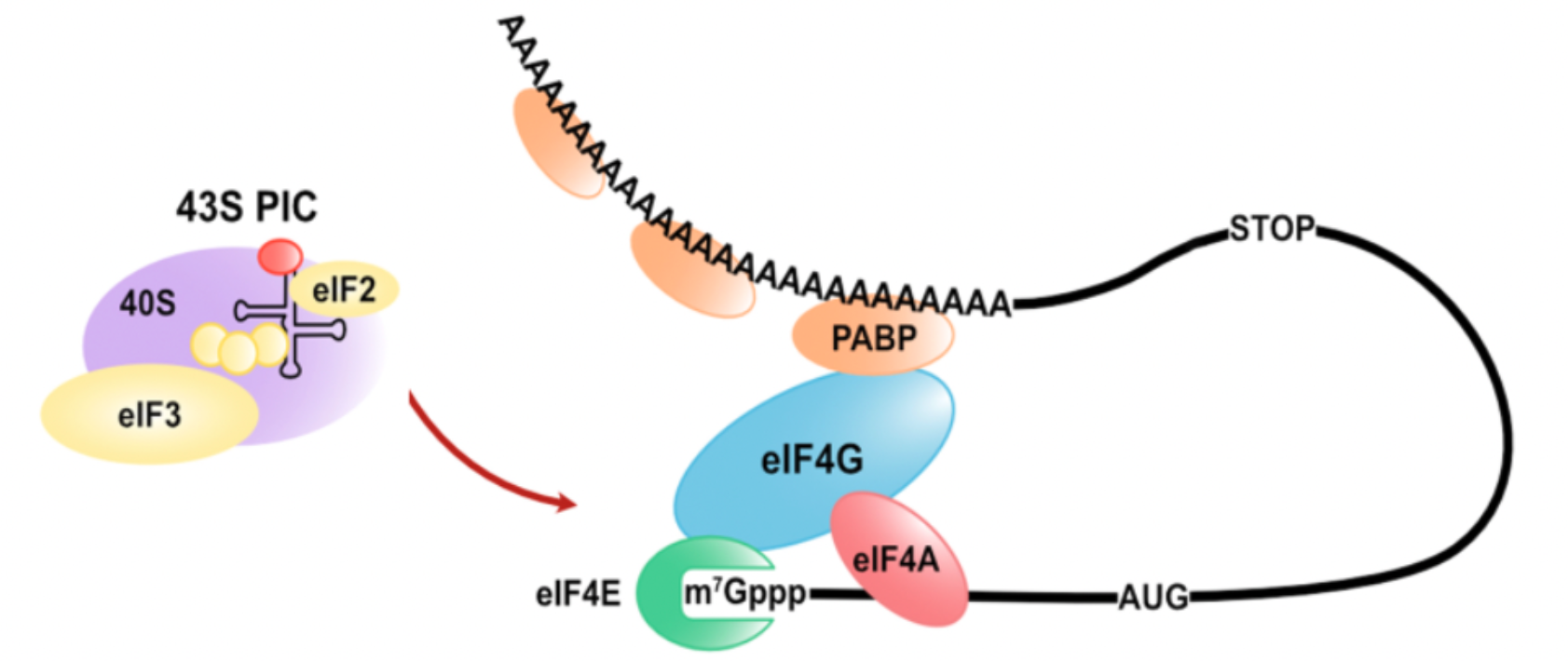
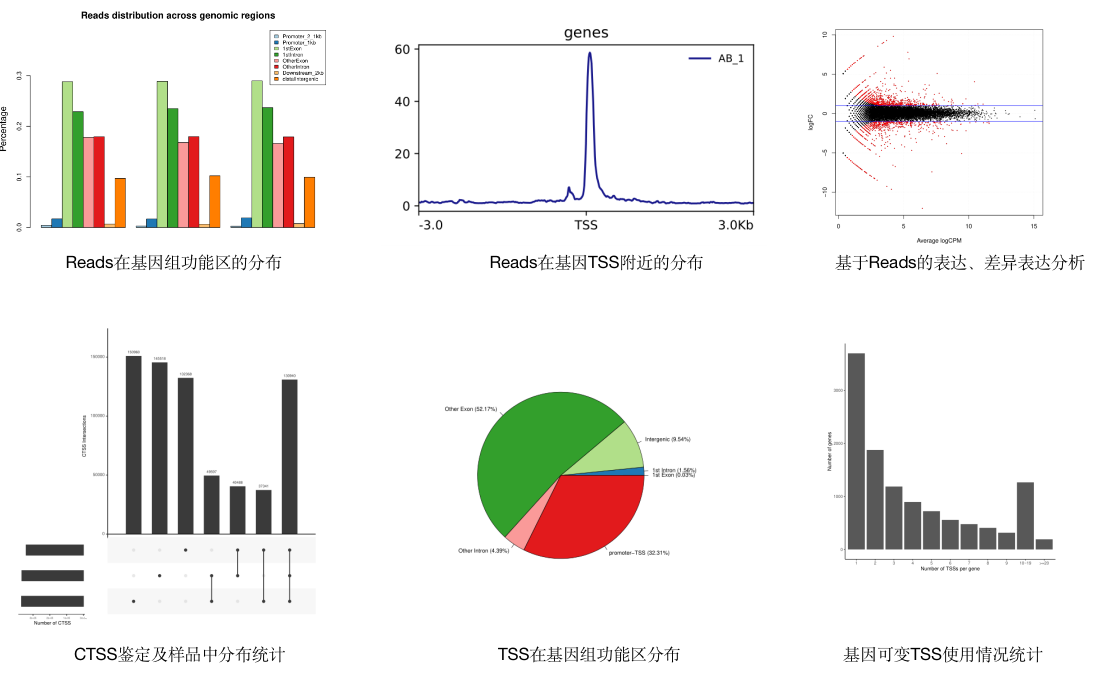
eIF4F复合物(包括eIF4A、eIF4E和eIF4G)识别mRNA的帽结构,并通过PABP的作用介导mRNA形成首尾相邻的环状结构,进而招募43S预起始核糖体复合物扫描AUG起始密码子,从而介导翻译的起始。同时,不同的5’ cap位点的使用,意味着基因使用了不同的转录起始位点(TSS)、不同的启动子,以及不同的转录因子的调控。因此对5‘ cap的研究具有重要的生物学价值。