20221209追击新热点:mRNA上的ac4C修饰,回顾真核生物翻译起始过程

#
引言
在mRNA转录后m6A修饰研究已经逐渐套路化后,ac4C修饰无疑为表观转录组学打开了新的大门,是目前研究的热点。ac4C修饰位点的鉴定主要通过抗体法acRIP-seq和化学法ac4C-seq技术来完成,关于这两种技术康测科技官网有详细介绍。由于ac4C在真核生物mRNA上修饰丰度比m6A低很多,因此acRIP-seq方法RNA起始量需求很高(>200 ug RNA)。另外一种技术化学法ac4C-seq虽然可以解决RNA起始量的问题(>30 ug RNA),而且实现单碱基分辨率,但是根据Nature主刊文献(Dynamic RNA acetylation revealed by quantitative cross-evolutionary mapping, DOI: 10.1038/s41586-020-2418-2)报道,化学法ac4C-seq在真核生物mRNA上鉴定到的位点极少(个位数),过表达NAT10(ac4C乙酰化酶)后位点数量达到百位数,但这与acRIP-seq鉴定出的peak数量(即ac4C修饰位点数量)相悖。本次文献来源于首次发现真核生物mRNA上ac4C修饰的实验室,他们修改了ac4C-seq化学处理时的化合物种类,将NaCNBH3替换为NaBH4(康测科技正在测试和评估),可以鉴定出与acRIP-seq相同数量级的位点,并且进行了ac4C修饰的功能研究,让我们赶紧看看这篇文章吧。
#
背景
该实验室前期研究通过acRIP-seq在HeLa细胞的mRNA中检测到数千个ac4C位点,其中大多数分布在CDS区。在化学机制上,ac4C的N4-乙酰基团产生一个分子内氢键并加强胞嘧啶核苷酸的构象,进而加强其与鸟嘌呤核苷酸配对的稳定性。因此,CDS区的ac4C修饰通过提高与同源tRNA的相互作用来增强翻译延伸过程,从而抑制可能发生在非最优密码子环境下的共翻译衰变途径。虽然CDS区ac4C修饰和翻译延伸之间的关联是明确的,但之前的数据并不能解释在ac4C修饰在5’UTR分布的偏好性,而且对整体mRNA表达水平没有影响。首先5’UTR区域能以多种方式与翻译机制相互作用,从而在翻译调控中发挥关键作用。
让我们回顾下真核生物翻译的过程:翻译起始因子通过与m7G帽子结构的相互作用聚集在mRNA的5’末端,并通过40S核糖体亚基与起始tRNA(tRNAi Met)结合,产生的前翻译起始复合物(PIC)沿3’端方向进行扫描直到遇到一个起始密码子(通常是 AUG),这是通过与 tRNAi Met反密码子环结合实现的。起始密码子识别元件允许60S核糖体大亚基加入并形成能够进入翻译延伸阶段的成熟的80S核糖体。除了为核糖体组装提供平台外,5’ UTR 元件对起始密码子的选择具有正面和负面的影响。如果围绕第一个AUG的核苷酸环境是不利的,并且缺乏保守的“ Kozak”序列,则核糖体可能组装失败。
作者围绕5’UTR区域的ac4C修饰,确定了其在翻译起始调控中的独特作用。在体外和体内实验中,5’UTR区域存在ac4C修饰的mRNA在预期位置上游的翻译起始增加,而在起始密码子处的翻译起始减少。ac4C修饰对其他最佳翻译起始环境的负面影响在机制上归因于碱基配对的改变,通过形成翻译起始抑制性结构和直接调控AUG侧翼Kozak序列中与C碱基产生有利的互作来实现。那么作者是如何一步步完成验证这些结论的呢?我们接下来看看正文部分。
#
研究结果
1. 5’UTR和CDS区域ac4C修饰对mRNA翻译存在相反影响
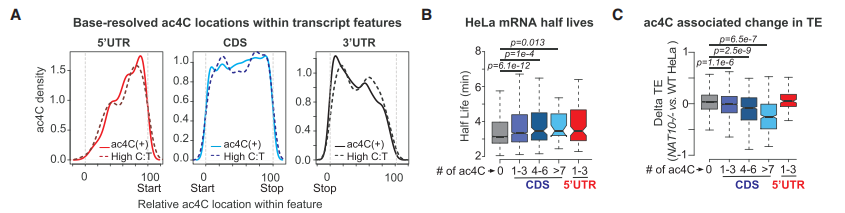
作者实验室之前通过acRIP-seq来鉴定发生ac4C修饰的区域,结果显示在野生型和利用CRISPR-Cas9敲除的NAT10的HeLa细胞中存在超过4,000个ac4C修饰区域,大多数发生在CDS区,通过促进翻译来提高mRNA稳定性,ac4C修饰在mRNA不同元件上的分布也表现出明显的5’端偏好性(下图A)。其中有22%的ac4C位点发生在5’UTR区域,但这些ac4C位点的缺失对mRNA的表达水平没有影响。同样,与不存在ac4C修饰区域[ac4C(-)]相比,CDS区存在ac4C修饰[ac4C(+)]区域通常会降低密码子的最优性,但是对于翻译起始位点(TIS)附近存在的ac4C修饰没有观察到这种趋势(下图A)。

为了评估ac4C修饰位于5’UTR和CDS区域是否会产生的不同影响,作者进行了体外mRNA翻译实验。在CTP或ac4-CTP存在下,体外转录萤光素酶mRNA均匀掺入CDS和 UTR区域。随后进行RRL(兔网状细胞裂解物)体外翻译实验,显示ac4C (-) mRNA萤光素酶活性快速增加并达到稳定的平台期 (下图B),而ac4C (+)荧光素酶活性初始滞后,接着出现强烈递增,且远高于ac4C (-)组,表明ac4C修饰同时具有抑制和激活翻译的作用(下图B)。
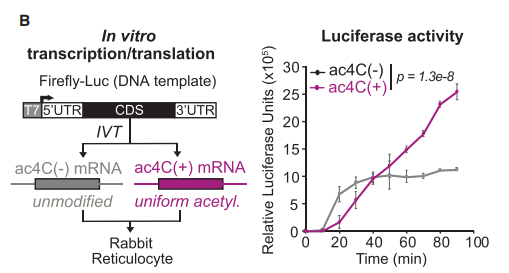
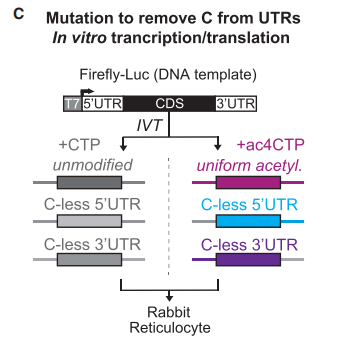
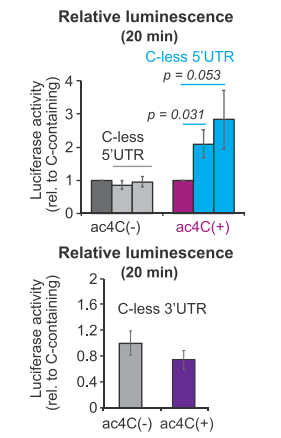
另外,通过突变去除所有5’UTR区的C碱基可以缓解ac4C (+) mRNA的萤光素酶产生的早期滞后,而ac4C (-)萤光素酶的翻译不受影响(下图C)。同样地,3’ UTR区C碱基的突变不会以ac4C依赖的方式影响萤光素酶的活性(下图C)。因此,mRNA翻译的早期抑制部分归因于5’UTR 中ac4C修饰的存在。
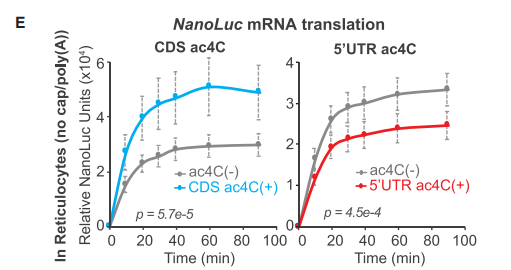
为了明确位置特异性的ac4C效应,作者通过Splint liagation产生纳米荧光素酶(NanoLuc) mRNA,可以在5’UTR或CDS区内指定位置引入ac4C修饰。通过RRL体外翻译发现TIS下游的六个串联密码子的ac4C修饰可以促进翻译过程,但在5’UTR区内加入相同的序列则会抑制蛋白质合成(下图E)。
2. RedaC:T-seq改良ac4C-seq
作者使用硼氢化钠(NaBH4)替代ac4C-seq中的NaCNBH3,命名为RedaC:T-seq(下图A),并从多方面对该方法的精确性、有效性及稳定性进行了评估。

首先相比ac4C-seq,RedaC:T-seq可以鉴定出更多ac4C修饰位点,比如WT HeLa细胞可以检测出7851个位点;而ac4C-seq根据康测经验,采用最宽松的参数分析只能鉴定出百位数的位点,如下图。
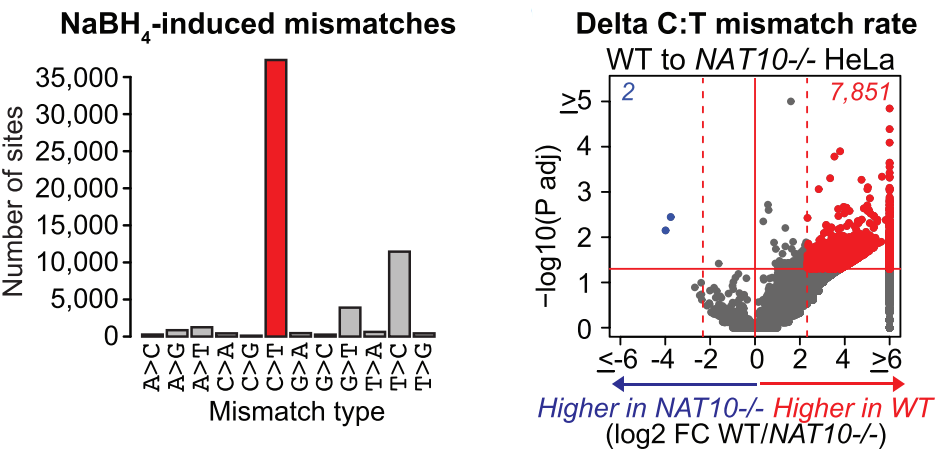
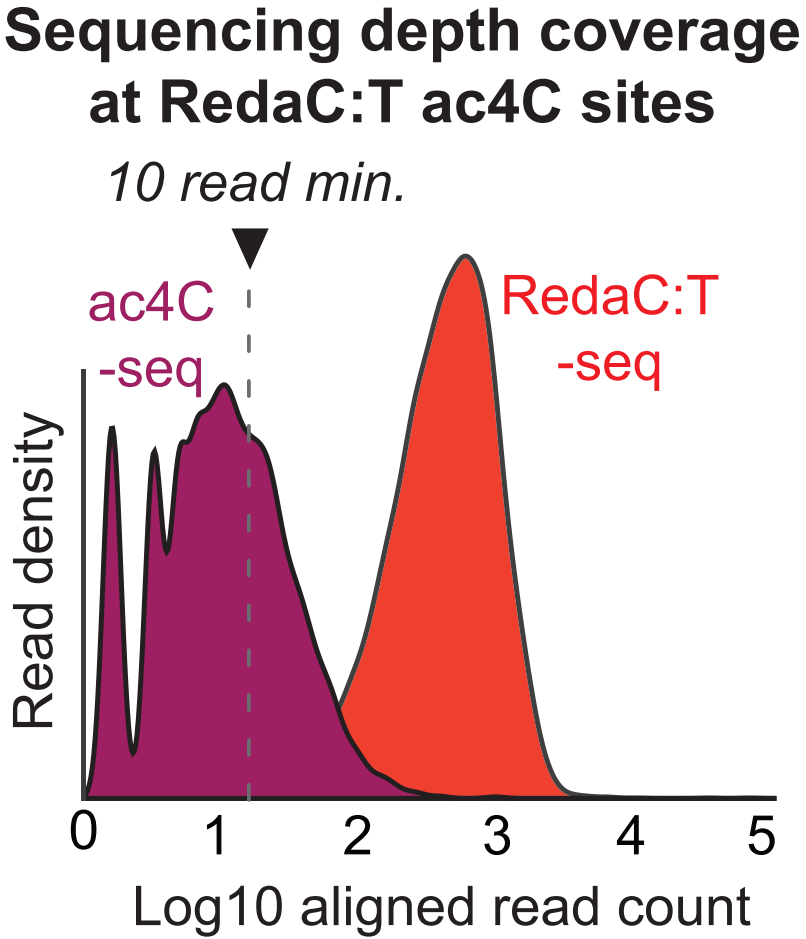
作者也分析了ac4C-seq数据,发现造成鉴定位点数量差异的原因主要是发生转换位点的覆盖深度有差异,ac4C-seq覆盖深度明显低于RedaC:T-seq,如下图。
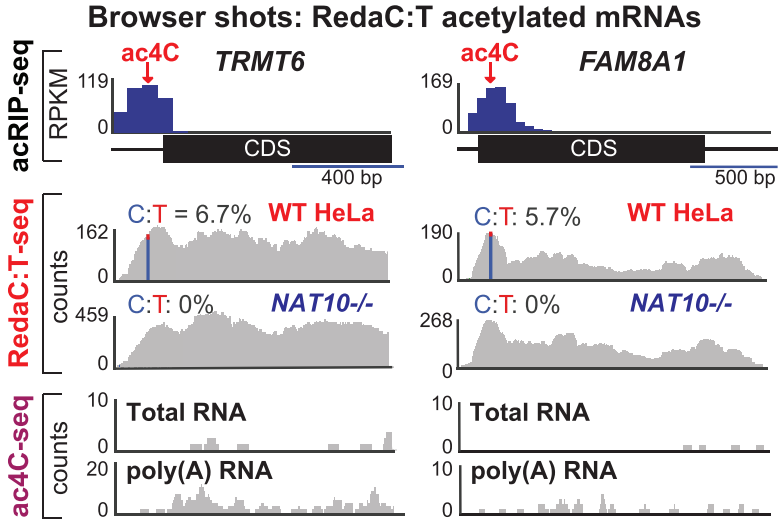
作者将自己的acRIP-seq、RedaC:T-seq和文献中报道的ac4C-seq放在一起进行比较,发现acRIP的peak和RedaC:T-seq鉴定的ac4C修饰位点位置吻合,而ac4C-seq并不能检测出该位点,如下图。
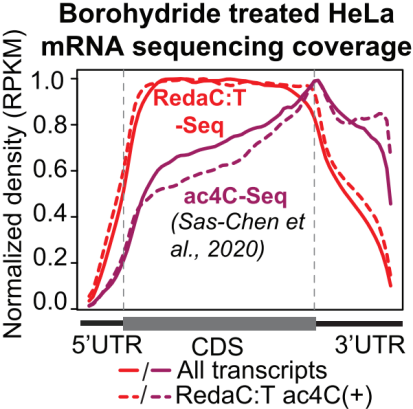
最后做出的ac4C修饰位点在mRNA各元件分布图也有差别,RedaC:T-seq在CDS5’端明显有更高的density,如下图。
3. 5’ UTR区ac4C修饰的存在可以增加上游翻译起始的比例
利用RedaC:T-seq技术,作者可以在体内确定ac4C修饰发生位置与mRNA翻译的关系,结果显示ac4C均匀分布在CDS区域中,而5’和3’UTR上的位点在翻译起始和终止密码子附近发生富集(下图A) 。RedaC:T-seq鉴定到的ac4C修饰位点与作者之前的acRIP-seq结果较为一致,如在ac4C修饰存在下可增强mRNA稳定性;而CDS区ac4C损失可以降低翻译效率(TE)。进一步分析发现随着mRNA的CDS区ac4C修饰位点数量增加,对NAT10缺失的敏感性增强(下图B,C),这说明通过敲除NAT10后5’UTR区ac4C修饰位点的损失对翻译效率没有影响。
为了强调5’UTR区ac4C位点对翻译起始过程的影响,作者进行在三尖杉酯碱(harringtonine, HR)存在的Ribo-seq。放线菌酮(CHX)可以结合在翻译过程中的E位点并阻止肽链延伸,而HR在加入40S小亚基之前通过结合60S大亚基中的A位点确定翻译起始的位置(下图D);与预期一致,结果显示HR reads在标注的翻译起始位点(aTIS)强烈富集,而CHX reads堆积在整个CDS区(下图E)。

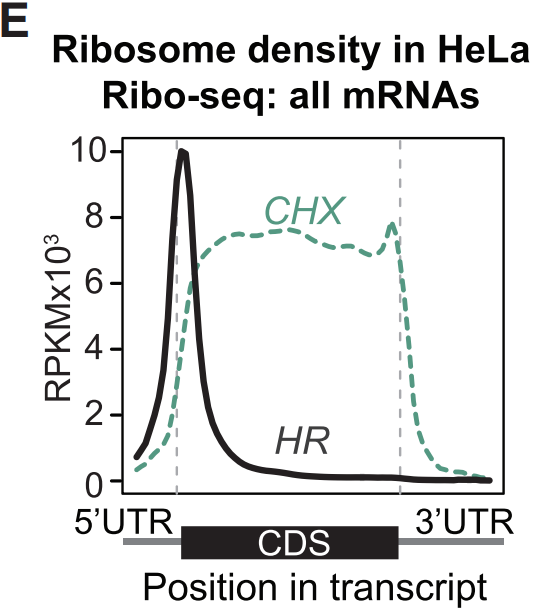
接下来作者根据ac4C修饰状态分离HR Ribo-seq结果,并检查aTIS周围的reads密度,结果显示与ac4C(-) mRNA相比,5’UTR区存在ac4C修饰的mRNA在aTIS上游reads发生显著堆积和预期翻译起始位点的相互减少。
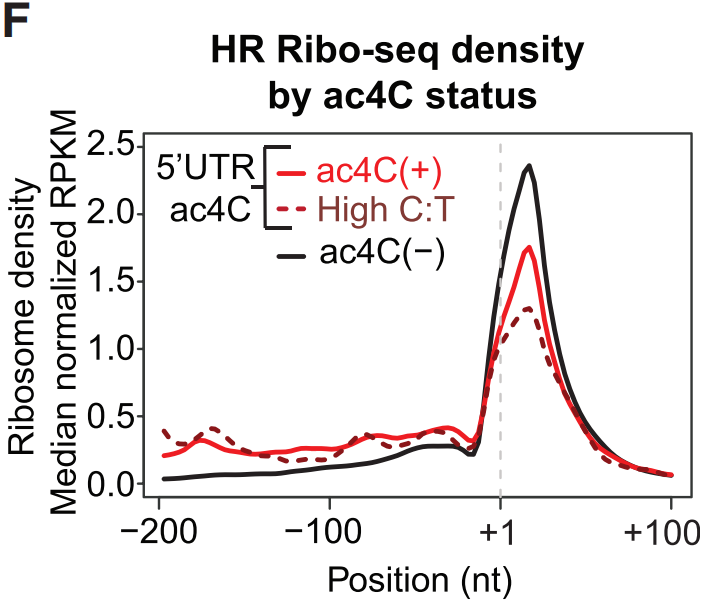
为了深入了解核糖体密度向5’位移的来源,作者检查每个5’UTR发生ac4C mRNA的HR Ribo-seq信号,发现大多数mRNA的主要起始位点发生在aTIS上游(下图H)。
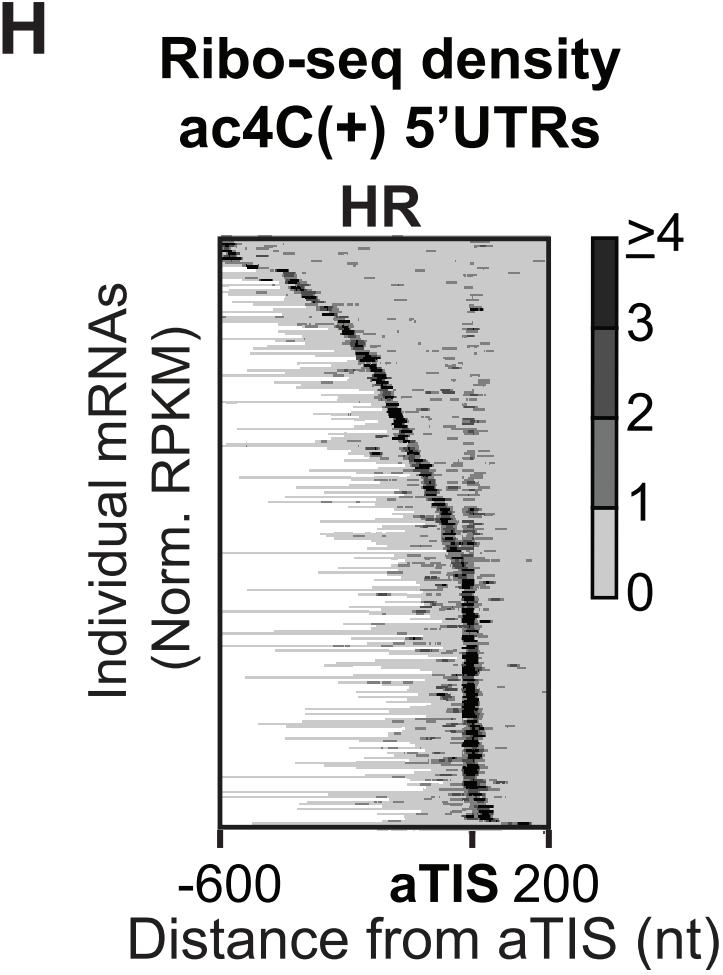
为了确认真实的翻译起始位点,作者分析CHX Ribo-seq结果中存在ac4C修饰的5’UTR区,结果显示与HR结果中排序的每个mRNA CHX Ribo-seq的热图显示了类似的富集模式,证实在HR Ribo-seq中检测到的起始核糖体的某些部分会继续主动进行翻译延伸。这些分析揭示了在5’ UTR区存在ac4C的情况下可导致非经典上游翻译起始的事件增多。
4. ac4C修饰直接抑制体外经典翻译起始过程
5’UTR区ac4C修饰与非典型起始同时出现,作者回归体外翻译研究ac4C修饰的功能。作者设计了如下实验(下图A)。

为了评估对上游翻译起始的间接影响,在与短ORF相关的非共识“弱”AUG密码子的下游区域引入ac4C修饰。NanoLuc翻译源自in-frame下游“aTIS” 的AUG,该AUG将受到upTIS翻译起始的竞争性限制(下图B)。考虑到ac4C促进mRNA高级结构形成,5’UTR高级结构通过改变可及性/核糖体扫描动力学影响翻译起始,作者进一步探讨高级结构对upTIS上ac4C功能的贡献,通过附近互补序列的引入(下图B)。在高级结构环境中,5’ UTR区ac4C修饰显著抑制NanoLuc的体外翻译过程,而ac4C修饰对无高级结构5’UTR的翻译没有影响。这些结果为非经典upTIS下游的ac4C修饰富集提供了解释,并确定核糖体扫描的阻塞是一种竞争性限制最佳下游aTIS获取的潜在机制。
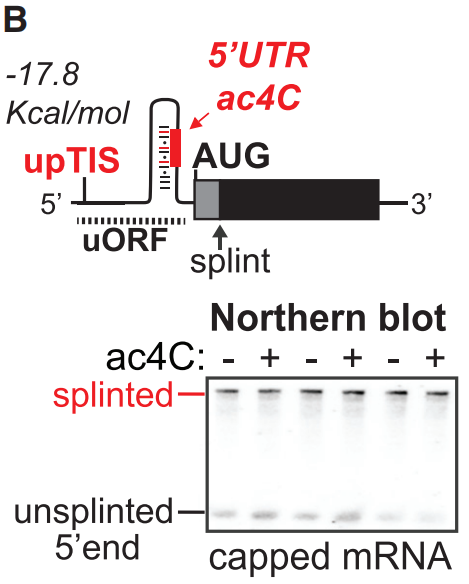

RedaC:T-seq和HR Ribo-seq联合分析进一步表明ac4C在起始密码子附近的作用,与无ac4C修饰的NanoLuc mRNA相比,Kozak序列中C碱基发生乙酰化可以显着抑制翻译(下图C)。总之,这些涉及具有位点特异性的ac4C修饰mRNA报告基因的分析最终确定在翻译起始中的作用。

5. 5’UTR区ac4C修饰调控体内翻译起始过程
结合体内核糖体定位和体外翻译实验的结果,作者揭示ac4C修饰在促进识别非最佳上游密码子同时抑制典型起始密码子AUG的双重作用。为了在细胞中验证该模型,作者研究5’UTR中ac4C修饰的损失是如何影响敲除NAT10条件下的翻译起始过程。起始核糖体在WT细胞中乙酰化5’UTR的mRNA分布与敲除NAT10的HeLa大致一样,如下图A。
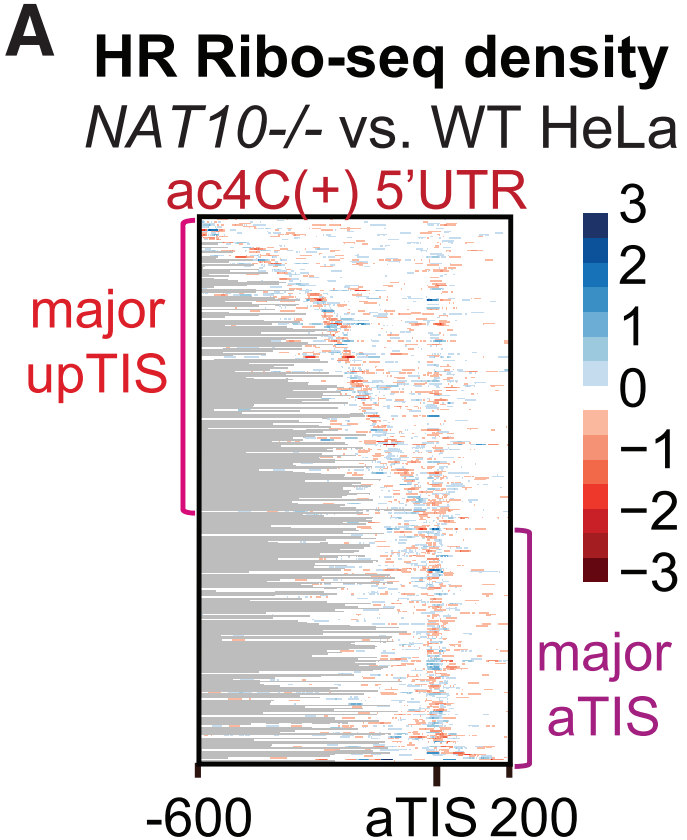
NAT10敲除可以轻微降低5’UTR区upTIS的ac4C修饰丰度(如下图B)。

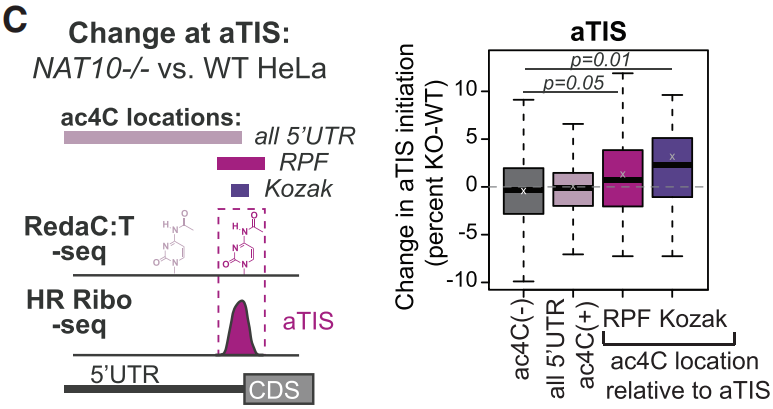
考虑到保守的Kozak序列富含C碱基,如果在Kozak序列C碱基出现ac4C修饰,其可以中和mRNA中AUG近端C碱基与扫描核糖体内组件之间的有利互作(下图C)。
编码paxillin(PXN)和角蛋白80 (KRT80)的mRNA具有5’UTR区ac4C修饰位点,去除该mRNA中5’UTR ac4C修饰可以导致核糖体密度向aTIS转移并增强蛋白质翻译能力,而5’UTR不存在ac4C修饰的GAPDH mRNA不受影响(下图D)。由ac4C促进的upTIS竞争性地限制aTIS的翻译起始,并且 ac4C 可以通过包含茎环结构的热力学稳定进一步阻塞起始密码子(下图E)。与WT HeLa相比,NAT10-/-细胞在划痕试验中显示增加的迁移(下图F)。
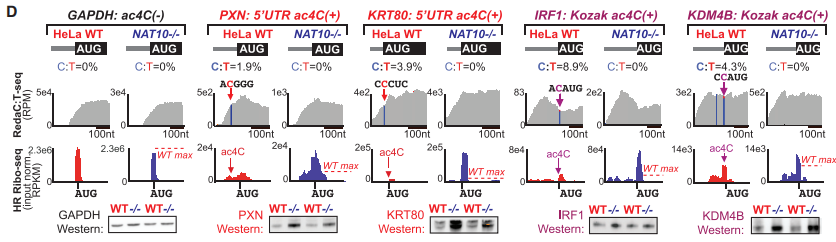
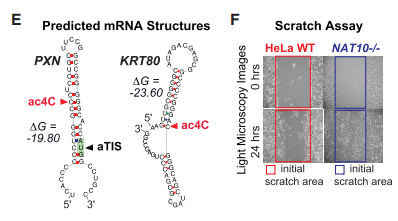
与Kozak序列中ac4C的抑制作用一致,转录因子IRF1和染色质修饰子KDM4B的AUG起始密码子的- 1位置的ac4C修饰缺失可以导致aTIS处核糖体密度大幅增加,蛋白翻译能力增强(上图D)。与细胞周期有关的KDM4B的mRNA也与IRF1一样在5’UTR区存在ac4C修饰,表明5’UTR区ac4C可能在调节基因表达和细胞生长中起作用(下图G)。

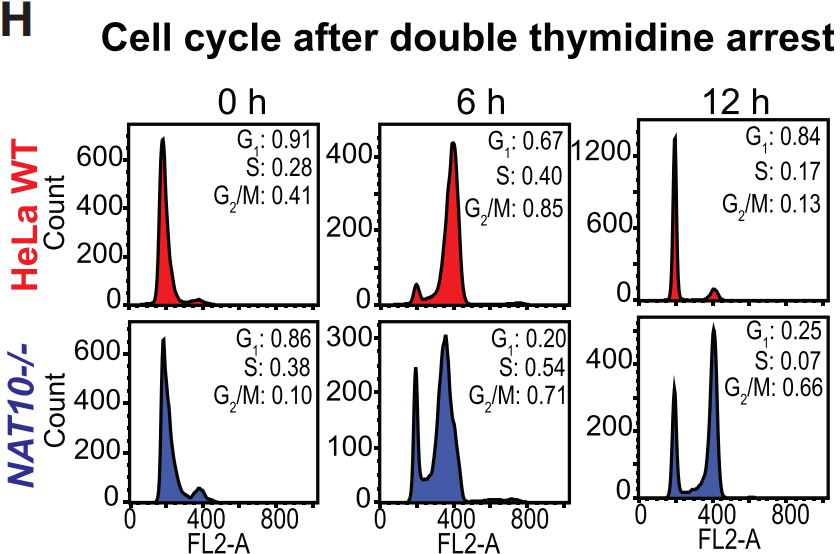
确实与WT HeLa细胞相比,敲除NAT10后的主要表型变化是细胞周期转变(下图H)。这些结果证明5’UTR区ac4C修饰在翻译调控中的作用而且暗示调控细胞行为是为了响应特定刺激。
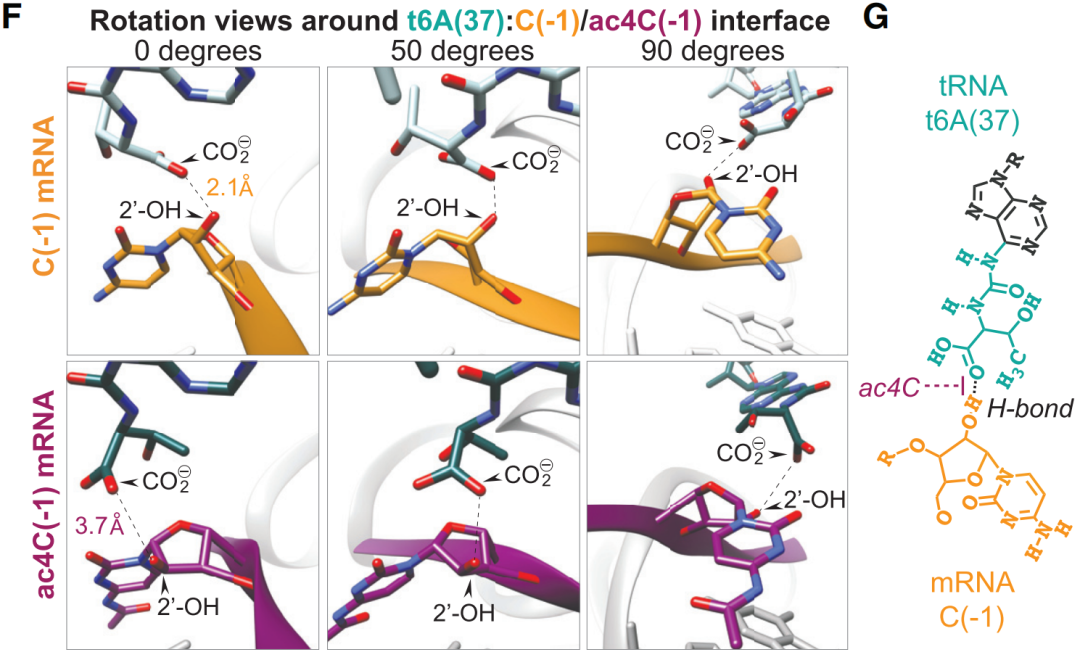
最后作者利用冷冻电镜技术阐明ac4C如果发生在Kozak序列内部时的翻译抑制作用机制:通过破坏tRNAiMet中与t6A的稳定相互作用。具体可以看原文献,这里不再展开。
#
总结
这篇文章深入研究mRNA 5’UTR区ac4C修饰是如何调控翻译起始的位点选择,主要有两大贡献:a. 开发RedaC:T-seq,使用NaBH4替换ac4C-seq的NaCNBH3能得到数千个ac4C修饰位点,与acRIP-seq的结果较为一致,弥补ac4C-seq鉴定位点较少的缺点;b. 确定5’UTR区ac4C修饰可以抑制翻译起始过程,机制是通过改变经典翻译起始位点至上游5’UTR区域并与经典AUG起始密码子竞争起始来完成的。
1. 通过体外翻译实验确定位于不同区域的(CDS/5’UTR)ac4C修饰对翻译过程有相反影响;
2. 通过HR Ribo-seq和RedaC:T-seq,发现5’UTR区如果存在ac4C修饰可以使经典起始密码子上游翻译起始的事件增多;
3. 再次通过体外翻译发现5’UTR区如果存在ac4C修饰会导致翻译起始受到抑制;进一步分析发现其机制是Kozak序列中C碱基发生ac4C修饰导致的。
4. 回到体内验证,在Kozak序列中添加ac4C修饰与体外结果一致:翻译起始受到抑制;在具有5’UTR区ac4C修饰特征的mRNA基因与细胞周期和转录调控有关。
5. 利用冷冻电镜阐明ac4C修饰发生在Kozak序列内部时抑制翻译起始的机制。
文献中康测已有产品:acRIP-seq、ac4C-seq
#
参考文献
Arango et al., Direct epitranscriptomic regulation of mammalian translation initiation through N4-acetylcytidine, Molecular Cell (2022).
DOI: 10.1016/j.molcel.2022.05.016











