20231201康测科技TCR-seq分析报告升级
“
康测科技TCR-seq分析报告升级
NEWS TODAY
T细胞受体(TCR)是T细胞表面的受体蛋白,其在识别和应对感染、病毒、肿瘤等生物威胁中起到关键作用。随着TCR-seq技术的不断发展,我们对T细胞免疫系统的理解得到了极大的拓展,为研究和开发新型治疗方法提供了宝贵的信息。目前,TCR-seq已经成为免疫学和临床研究中不可或缺的工具之一,对于免疫疾病治疗和癌症免疫疗法的研究具有重要的意义。
康测科技作为国内最早提供TCR-seq测序服务的供应商之一,凭借基于RNA检测的5’ RACE方法,一方面可以获得全长TCR信息,更重要的是避免了基于DNA检测的V-J引物偏好性的问题,结果更加稳定、定量更加准确。康测科技的独门秘笈UMI去重技术,进一步提高了结果的准确性;UMI的纠错功能,又可以过滤掉建库过程中引入的假阳性CDR。综合上述特点,康测科技TCR-seq的准确性、真实性、稳定性、一致性一枝独秀,为客户提供可靠的检测结果。已支持客户在Cancer Cell、American Journal of Transplantation等权威杂志发表了重量级研究成果。
作为测序行业的技术咖,对技术的更新和对产品的打磨,一直是康测科技不懈的追求。为了更好地满足日益提高的研究需求,我们对现有的TCR-seq分析报告进行了升级。这一升级旨在为相关研究提供更全面、更多样的TCR-seq分析内容,以更好地支持科学家和医学研究人员的工作。
本次报告升级主要优化与新增以下分析内容:
“
1.更完善的数据库注释
/ NEWS TODAY
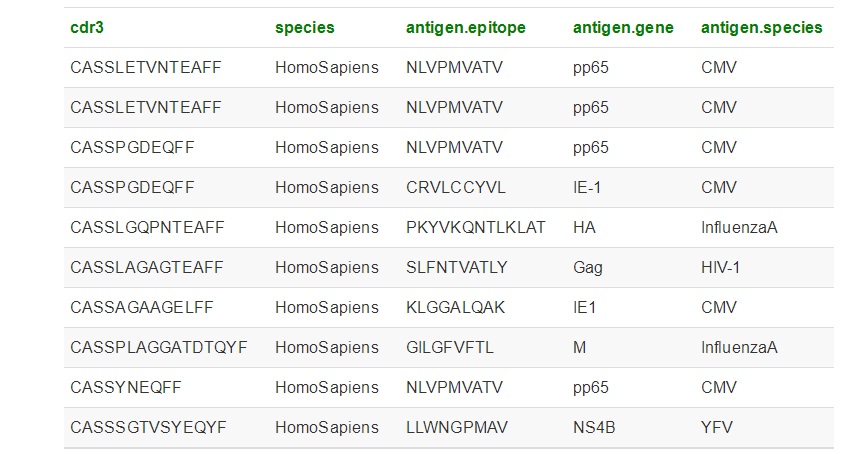
由于TCR序列庞大的多样性以及缺乏对T细胞特异性大规模数据的整理,TCR序列的注释,尤其是涉及抗原的相关注释,一直是一个具有挑战性的任务。VDJdb数据库存储并整合了已发表的T细胞特异性分析结果,为将抗原特异性与TCR序列结合提供了通用平台,可用于TCR序列数据的通用注释(Shugay et al. 2018)。
在本次报告升级中,我们使用最新版本的VDJdb作为背景数据库,对样本的CDR3区段进行详尽的注释,以预测可能的CDR3序列结合的抗原肽段。这有助于确定在一系列免疫介导条件中样本之间具有差异的CDR3序列,并进一步确认其为潜在的CDR3序列。相较于直接通过某种克隆型的丰度变化来筛选,通过关联的抗原注释,我们能够更清晰地发现多个具有相同或高度相似的氨基酸序列的TCR受体。此外,对于已知抗原的研究,也能更有针对性地选定对应CDR3序列。
这一升级将为研究人员提供更全面、更准确的TCR序列注释,使其能够更深入地理解T细胞免疫反应的抗原特异性,为个性化治疗和疾病机制的研究提供更为精准的基础。
“
2.更多样的可视化方式
/ NEWS TODAY
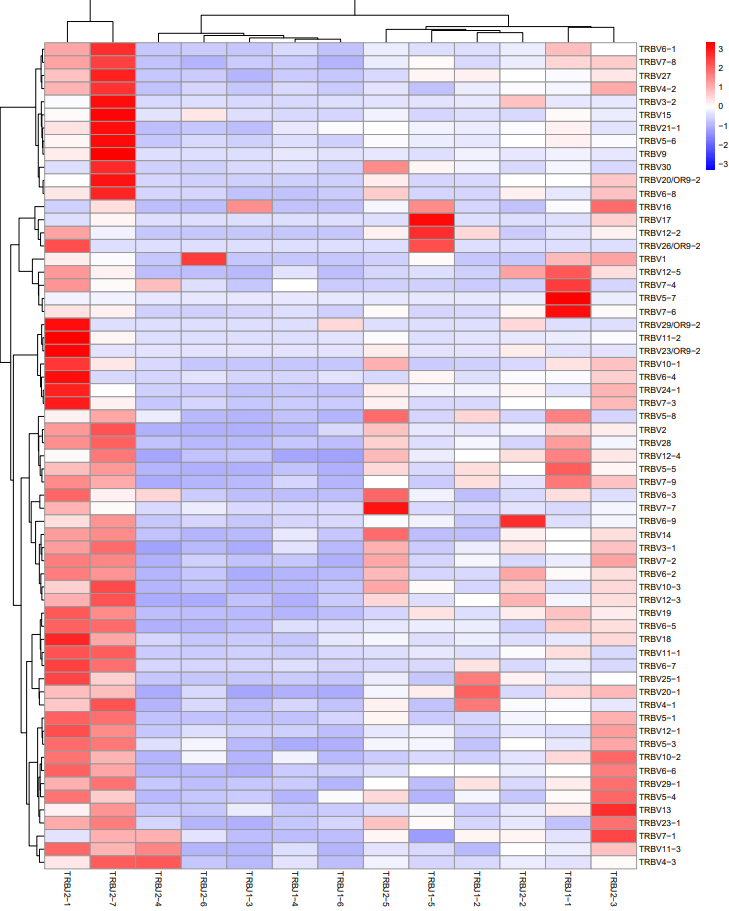
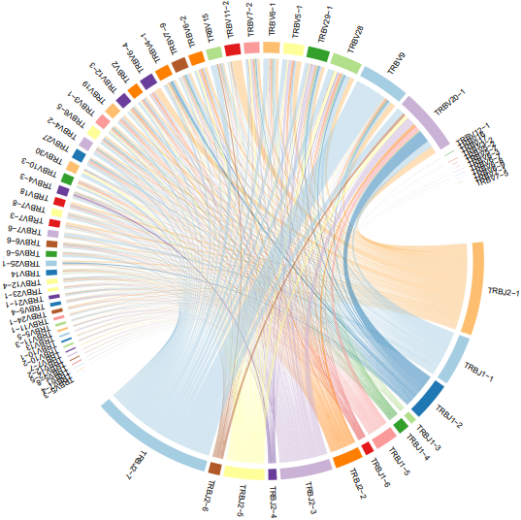
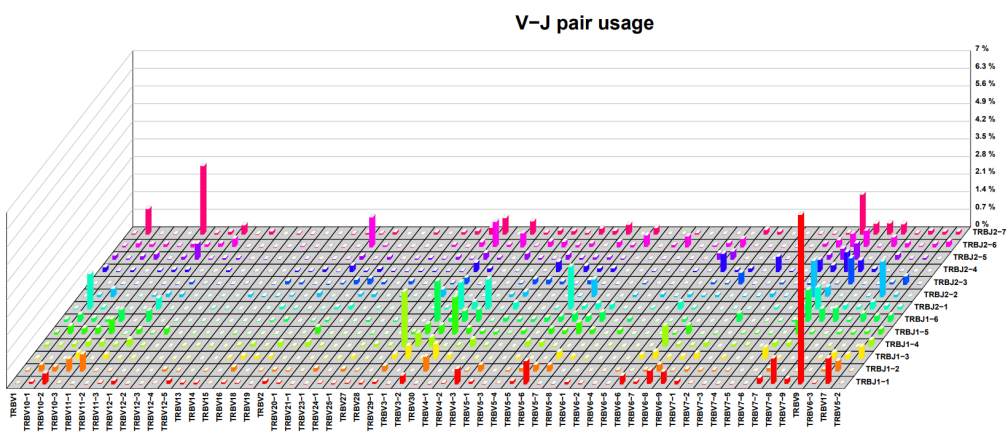
在此次报告的升级中,我们充分整合了当前文献中广泛采用的多种结果可视化方式,来更全面地呈现TCR-seq数据的复杂信息。除了提供常规的森林图来直观地展示VJ基因的频率分布之外,本次更新引入了热图和弦图,从而更加生动地展示VJ基因之间的关联关系,以及VJ-pair在样品间的量变,为用户提供更详细的TCR序列变化图谱,使用户更容易理解不同条件下的TCR序列的动态变化。
这些多样化的可视化方式有助于研究人员更深入地挖掘数据,从而更全面地理解T细胞免疫系统的动态变化。通过提供这些清晰、生动的结果展示,我们旨在使用户能够更轻松地解读复杂的TCR-seq数据,并更有针对性地指导后续的实验设计和研究方向。
“
3.更全面的多样性指数统计
/ NEWS TODAY
TCR种类理论上可达1016-1018种,也因此在分析中使用量化指标是必不可少的,以便评估TCR的多样性,从而反映克隆的不同类型数量,并同时考虑这些类型的个体分布之间的系统性关系,包括但不限于丰富性、差异性和均匀性等。
在本次报告升级中,我们引入了一系列当前TCR研究文献中常用的多样性指数统计指标。与之前版本中简单的香农(Shannon)、辛普森(Simpson)和D50指数统计相比,我们提供了更为全面的多样性指数统计,包括:
1.Clonality克隆性指数,用以评估TCR克隆的扩增情况。Clonality值越大,表示有部分TCR克隆发生了很大频率的扩增。
2.Pielou指数,用以评估克隆分布均匀程度,Pielou值越高,表示各克隆分布比较均匀, Pielou值越低,表示有一些特异性的克隆发生了扩增。
3.Chao1指数,用以评估克隆丰富度,Chao1值越高,表示克隆型种类越丰富。
上述指数的引入旨在帮助用户更好、更全面地评估样品的TCR多样性状况。这些指标涵盖了更广泛的方面,使用户能够更准确地了解T细胞免疫系统中的多样性,为深入的研究和临床应用提供更全面的数据支持。

“
4.更详尽的克隆性分析
/ NEWS TODAY
TCR克隆性分析在深入了解免疫系统在疾病、治疗和免疫应答中的作用方面具有至关重要的意义。这些分析有助于揭示T细胞克隆的多样性、动态变化和功能,为个性化医学、疫苗研发和免疫治疗提供有力的支持。
为此,我们在报告升级中引入了克隆性分析,并采用了三种不同的方法对克隆性结果进行分析与可视化展示,分别为占比最高克隆组(Top)、占比最低克隆组(Rare)、以及特定丰度所占比例的克隆组(Homeo)。这一改进相较于之前仅对Top100的克隆型进行频率分布统计,为用户提供了更全面、直观的不同样本间T细胞克隆的多样性、动态变化等信息的展示形式。
这些结果不仅展示了克隆组在免疫组库中的相对比例,还提供了关于特定丰度克隆组的详细信息,从而能够更全面地了解T细胞克隆的特征。
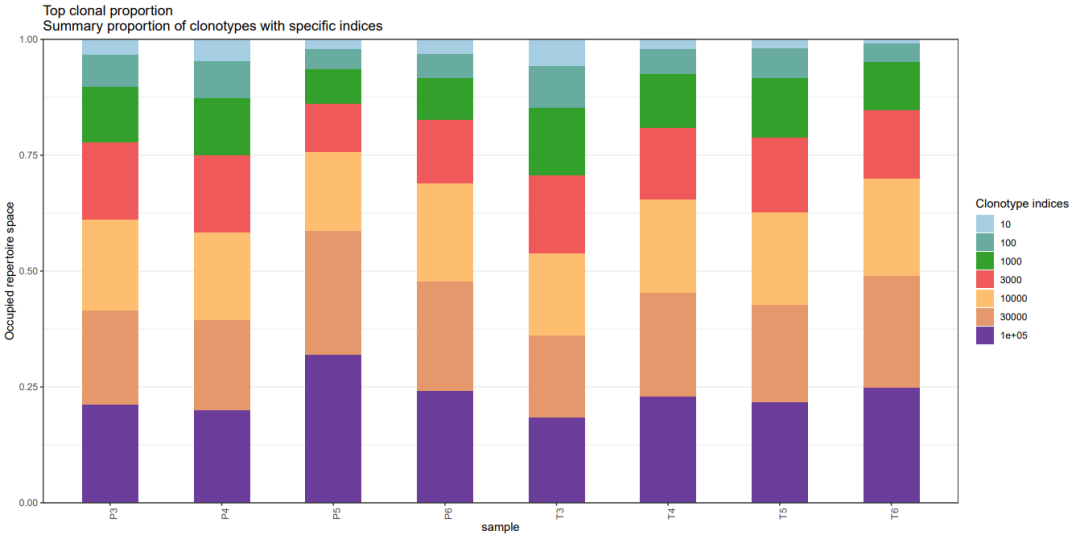
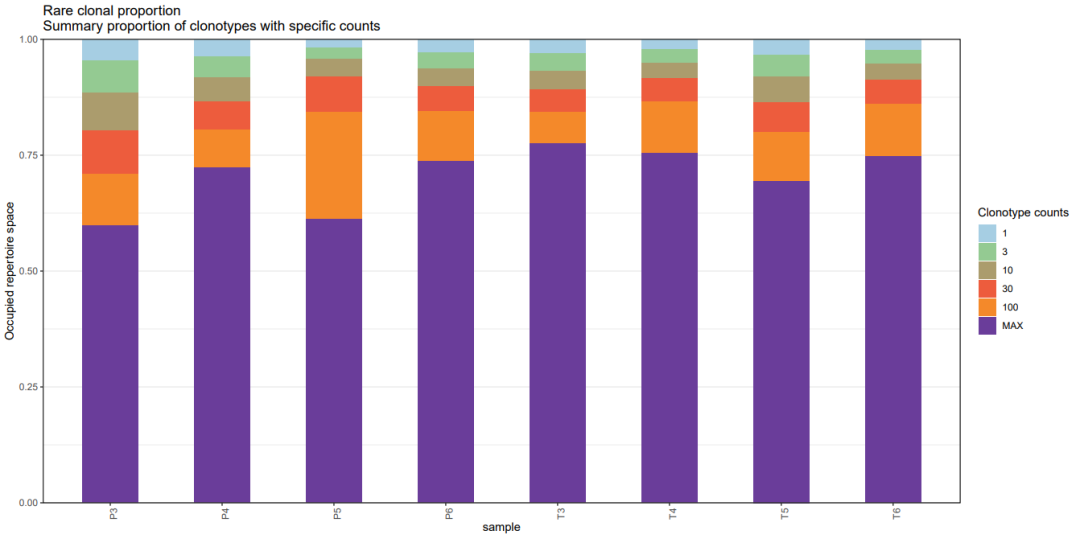

“
5.更丰富的共有克隆分析
/ NEWS TODAY
共有克隆分析是研究不同样本或个体中存在的相同T细胞克隆的关键环节,其目的在于识别在样本间共同存在的T细胞克隆,从而深入了解T细胞克隆的共性和个体差异,为免疫学研究、临床诊断和治疗提供重要信息。
在本次报告中,我们进一步丰富了共有克隆分析的内容。除了基本的表格和相关热图结果统计外,我们还引入了多样本间独有及共享克隆的venn图和upset图的可视化展示方案。这一改进不仅让用户能够更全面地评估样品间的相似性,还能够清晰地识别样品组内和组间共有的克隆类型。这些图形展示方案为研究人员提供了更直观的工具,有助于发现潜在的生物学模式和样本特征,为进一步的研究和治疗决策提供更多信息。
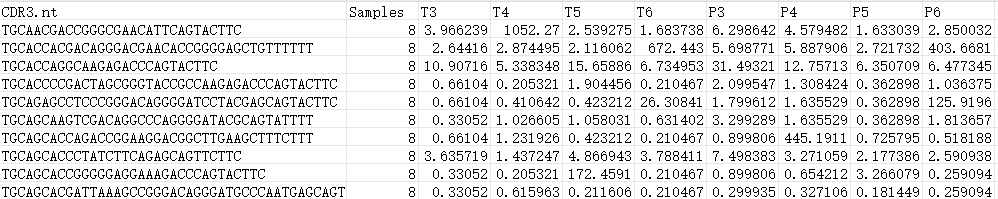
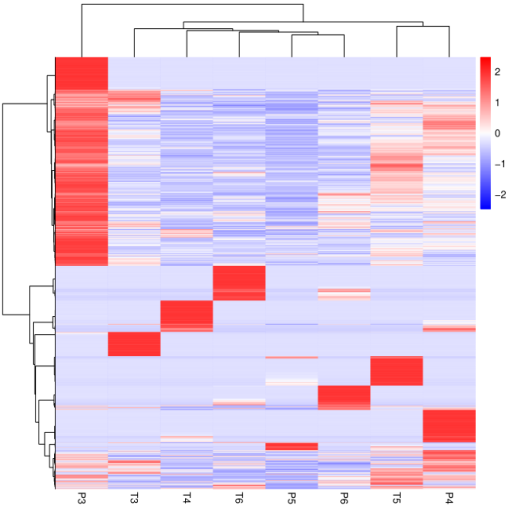

“
6.更有力的差异分析
/ NEWS TODAY
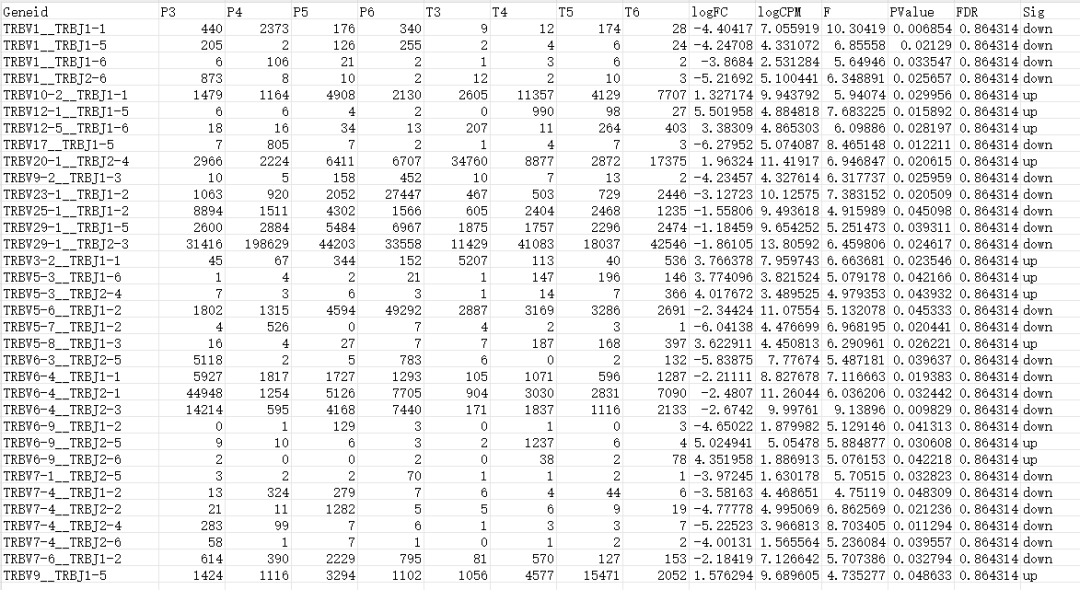
差异分析是科学研究和数据分析中的经典方法,为深入了解数据集和研究问题提供了基础。在TCR分析中,共有克隆分析虽然为初步探索组内/组间TCR的组成提供了基本了解,但为了确定其中的特征性克隆往往需要更进一步的数据挖掘。
在本次报告升级中,我们引入了对不同组别TCR克隆对应的VJ基因对和CDR3序列的差异分析。这一改进使用户能够更好地筛选出在不同组别之间存在显著差异的TCR克隆,为后续研究提供有针对性的候选集。通过对VJ基因对及CDR3序列的差异性分析,用户可以更清晰地了解在不同实验条件或个体之间的TCR变化,为深入挖掘潜在生物学模式提供更有力的支持。
这一差异分析的升级旨在帮助用户更精确地确定组间的特征性TCR克隆,为免疫学研究和治疗策略的制定提供更深入的洞察。

康测科技一直致力于不断提升产品和服务质量,以满足客户的需求。此次的TCR-seq报告升级旨在使用户更轻松地进行深入的TCR-seq分析,我们期待着新的升级能够为您在研究或医学实践中取得更多成就。如果您需要更多信息或示例报告,欢迎随时联系我们。感谢您对康测科技的支持,我们期待为您提供更优质的服务和支持。

康测科技
电话:027-65527552
网址:www.seqhealth.cn











