仅用10mg果蝇睾丸样本,能做好ATAC吗?(珍贵/微量样本值得一看)
仅用10mg果蝇睾丸样本,能做好ATAC吗?(珍贵/微量样本值得一看)
引言
ATAC-seq(Assay for transposase-accessible chromatin sequencing)是目前染色质构象研究的主流工具。其原理是,通过Tn5转座酶在细胞核内的染色质DNA上进行随机转座:
1.开放区域的染色质由于DNA暴露,因此可以发生转座;
2.紧缩区域的染色质由于DNA被压缩折叠,转座酶无法接近,因此不能发生转座。
转座成功的DNA被加上测序用接头序列,通过PCR扩增、高通量测序和生物信息学分析,染色质开放区域会产生测序reads,而染色质紧缩区域则无测序reads产生:染色质开放程度越高,其测序信号越强。
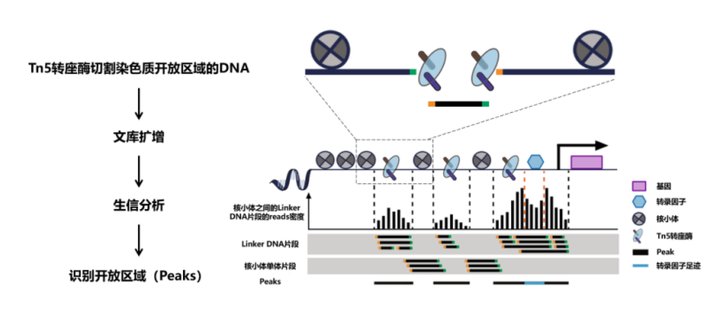
我们都知道ATAC-seq在微量组织中的应用是比较困难的,但困难不等于做不了,今天小编就给大家带来10mg果蝇睾丸组织的ATAC-seq~
物种:黑腹果蝇
组织:睾丸
样本量:10mg左右(常规ATAC需要100mg样本,该样本属于微量样本)
细胞核抽提结果展示
荧光计数:

提核后用AIPO染色,发现:
1.荧光信号呈现规则圆形;
2.荧光背景干净,说明细胞核纯度较高,杂质较少,更有利于后续实验。
建库结果

核小体单体条带清晰明显,说明建库成功
测序质控

文库插入片段长度分布:
测序文库插入片段大小的分布可以用来判断ATAC-seq实验的质量。由于Tn5转座酶优先攻击染色质开放区,在实验条件下会得到各个长度梯度的gDNA片段,一般来说,大多数DNA都是核小体自由区片段(Nucleosome-Free Regions fragments,<100 bp)或只包含一个核小体(~200 bp)的短片段,同时也有一些包含多个核小体的长片段,在含量分布上会呈现明显的片段分布特征。上图也可看到特征峰在<100 bp和接近200bp处特征明显,与前期实验结果一致性良好。

Reads在TSS附近的分布
以比对后得到的唯一比对序列为分析对象,分析其在基因TSS附近的深度分布,得到以上区间深度分布结果。由于NFR fragments应该富集在转录起始位点(TSS)附近,所以 ATAC-seq实验成功应在图中 TSS 处有明显的富集峰,富集越明显表示实验效果越好。
联系我们
地址:武汉市江夏区东湖新技术开发区高新六路99号南山·光谷自贸港A栋6楼
邮箱:seqhealth@seqhealth.cn
电话:027-65527552
热门服务
关注康测公众号













