-
-
-
核心技术 >
检测服务 >
产品/解决方案>
-
Direct RNA-seq
细胞的转录组包含丰富的信息,包括基因结构(如剪接变异和融合基因)、转录本的不同表达水平、反义转录,以及RNA上的表观修饰。基于Illumina平台等二代cDNA测序、ONT和Pacbio平台的三代全长cDNA测序已经很全面地描绘了转录组景观,但仍然存在一定的问题。比如二代测序平台采用Oligod(dT) 逆转录全长cDNA,或将RNA片段化后用随机引物逆转录成cDNA,最后使用PCR扩增出文库进行测序;三代测序平台比如Nanopore同样采用Oligod(dT) 逆转录全长cDNA,PCR扩增出文库后测序。由于这些方法中,cDNA经过PCR 扩增,这可能会引起扩增偏好性、降低cDNA文库的复杂度、干扰表达丰度定量以及影响某些RNA类型的检出。
有没有一种可以准确、区分链特异性,能够同时鉴定RNA修饰碱基的种类,而且不必担心在文库制备时PCR造成的伪影的方法?Nanopore Direct RNA-seq可以满足您对转录组的一切想象。
Direct RNA-seq测序原理
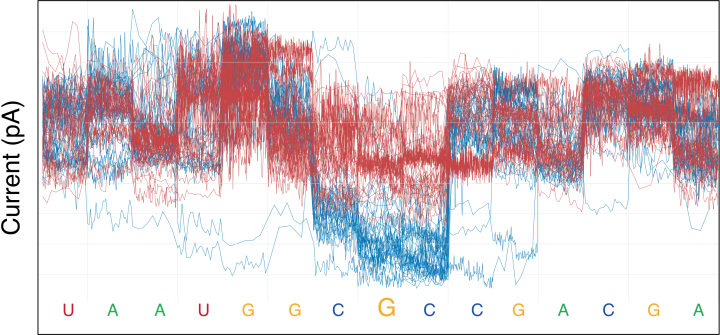
Nanopore Direct RNA-seq的基本原理是:单个DNA分子首先在施加的电势下进入纳米孔,然后通过解旋酶马达调控天然RNA的运动速度,使其逐个碱基地通过孔。在此过程中,流经孔的离子电流取决于流经孔的碱基序列,通过在大量RNA测序数据上训练的神经网络将这种离子电流特征转换成RNA序列(下图左),同时通过比较具有修饰和未修饰的RNA碱基电流信号的不同,或统计发生修饰位置错配率明显升高,就可以鉴定相应的RNA修饰(下图右,蓝色曲线代表存在修饰的RNA)。
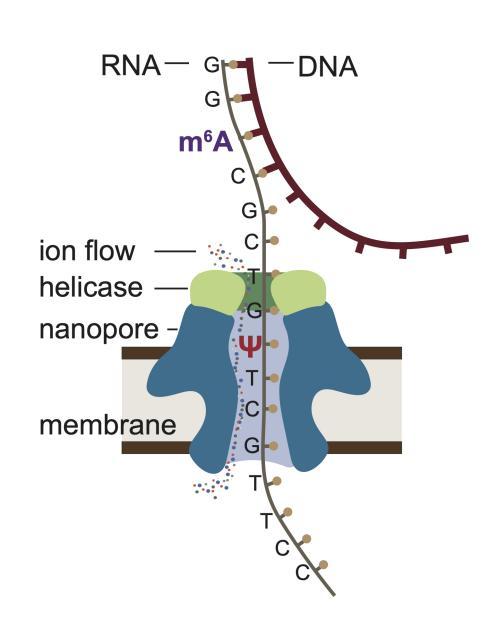
Direct RNA-seq建库原理
Direct RNA-seq建库步骤主要有:
- PolyA捕获富集Poly(A)+ RNA
- 利用Oligo dT逆转录成cDNA第一链(RNA起始量要求高,测序时间更长,数据量产出更高);
- Direct RNA测序;

Direct RNA-seq应用
鉴定RNA修饰
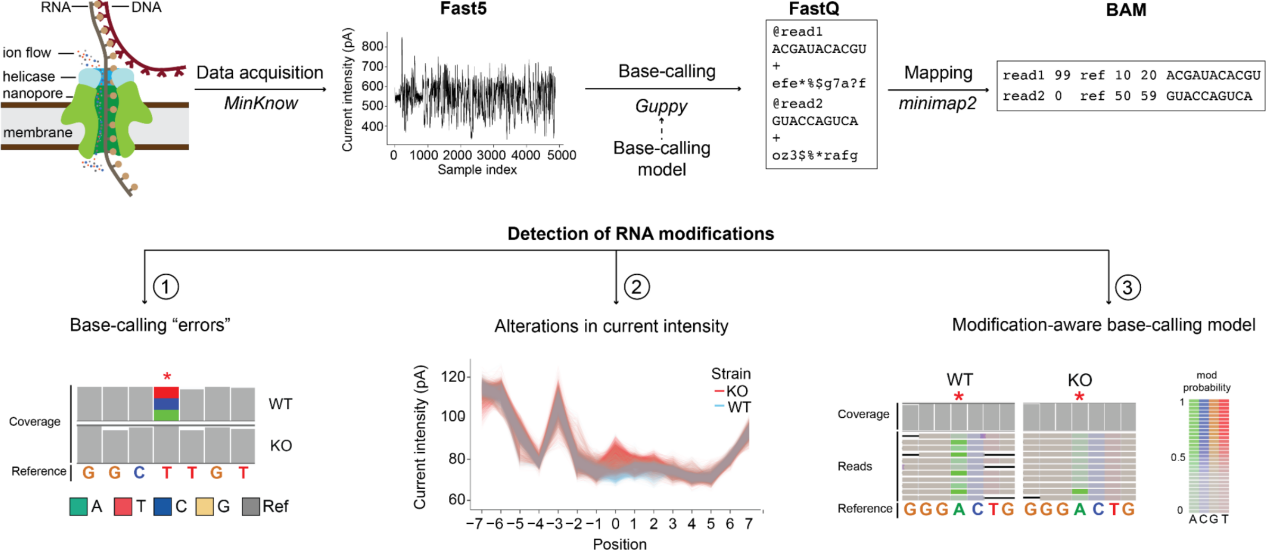
DRS鉴定RNA修饰的流程:
- MinKNOW软件对电流强度信息进行采集和处理,生成Fast5格式文件;
- Guppy等软件对Fast5进行Base-calling生成FastQ格式文件;
- FastQ文件可以通过比对软件如minimap2进行参考基因组比对,生成bam文件。
- DRS RNA修饰鉴定目前有三种策略:
- 以非随机base-calling错配的形式识别RNA修饰,表现为修饰位点相比上下游位置Base-calling错误率增加(下图①);
- 对比有修饰和无修饰位置的原始电流强度(信号强度、停留时间或迹线特征)来识别RNA修饰(下图②);
- 在进行Base-calling步骤时,使用感知修饰(Modification-aware)Base-calling模型,而不使用预测4种碱基的常规Base-calling模型。
目前已经开发出很多软件用于DRS的RNA修饰鉴定,如下表,康测m6A修饰检测使用的是MINES软件。


- 康测DRS实测数据(展示m6A修饰在RNA功能区分布)B. 康测DRS实测m6A保守motif
鉴定可变剪接种类
与Nanopore cDNA测序一致,DRS也可以鉴定基因的可变剪接,以及定量其产生的转录本。


康测实测可变剪接种类统计
*SE:外显子跳跃;MX:互斥外显子;A5:5’端可变剪接位点;A3:3’端可变剪接位点;
RI:内含子保留;AF:第一个外显子可变选择;AL:最后一个外显子可变选择
鉴定PolyA长度和APA位点
DRS除了可以鉴定各种RNA修饰、可变剪接种类以外,还可以直接鉴定APA位点和PolyA尾长度,如下图。
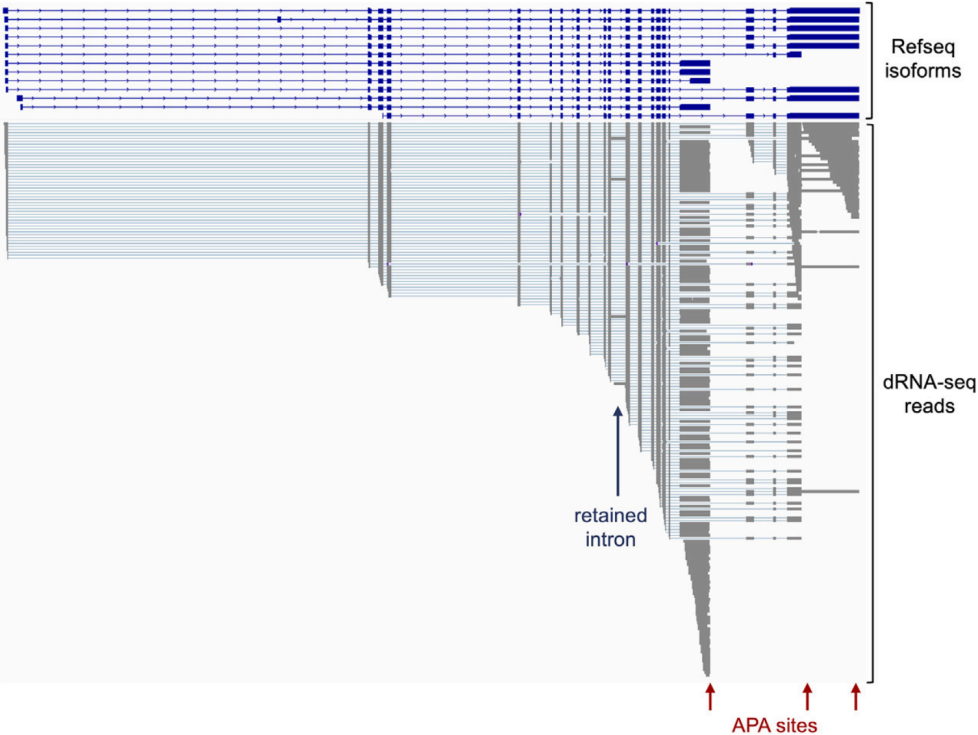

康测实测PolyA长度分析
Direct RNA-seq RNA修饰文献案例


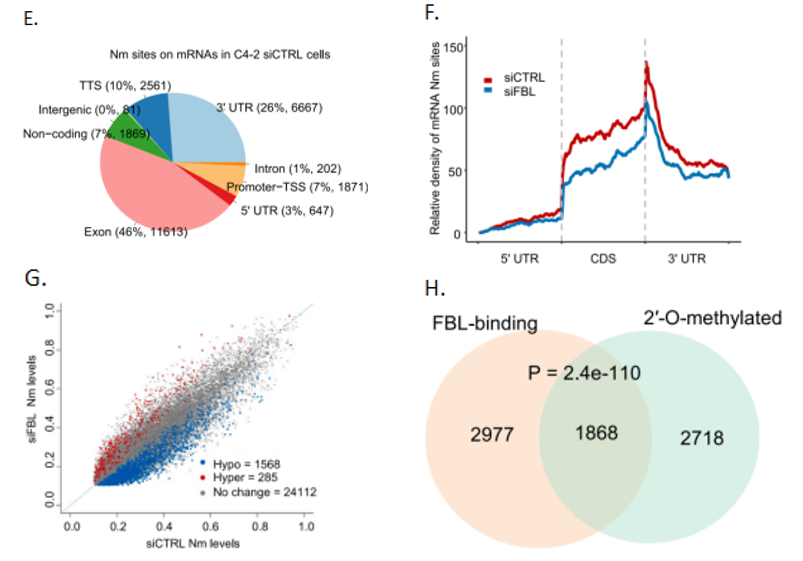

2'-O-甲基化(2'-O-Methylation,简称Nm)是一种特殊的核苷酸修饰,它能够作用于包括腺嘌呤(A)、尿嘧啶(U)、胞嘧啶(C)和鸟嘌呤 (G)在内的多种核苷酸。Fibrillarin(FBL)是一种重要的癌基因,特别是在TP53基因发生突变后,FBL会被异常激活。它的激活不仅促进了rRNA的2'-O-甲基化修饰,还加快了癌蛋白的翻译速度,从而参与了前列腺癌以及其他多种肿瘤的发展过程。本研究的结果包括:
- 公共数据寻找mRNA内部Nm修饰与稳定性的关联——内部Nm修饰增加mRNA稳定性(图A);
- 沉默FBL和配体NOP56进行RNA-seq验证表型——没有Nm修饰,mRNA半衰期降低(图B);
- Nm修饰的writer蛋白FBL进行RIP-seq鉴定其结合靶标,发现FBL未结合的RNA表达水平降低(图C);
- Direct RNA-seq鉴定Nm修饰位点,开发Nm修饰检测软件,准确鉴定Nm修饰(图D);
- Nm修饰在mRNA内部主要定位在Exon和3’UTR(图E);
- 沉默FBL可以全局性降低mRNA的内部Nm修饰水平,差异甲基化分析发现整个转录本Nm修饰均匀下降(图F和图G);
- RIP-seq联合DRS的Venn图鉴定FBL调控的靶标,表达水平越高的基因,其具有Nm修饰的mRNA比例越高(图H和图I);
- 寻找Nm影响RNA稳定性的机制:具有Nm修饰的mRNA 3’UTR长度更短,敲低FBL后3’UTR长度变长(图J);
- Nm可能影响可变PolyA位点选择,缩短3’UTR长度,从而增加mRNA的稳定性(图K)。
DOI: 10.1016/j.molcel.2024.04.011
Direct RNA-seq
联系我们
地址:武汉市江夏区东湖新技术开发区高新六路99号南山·光谷自贸港A栋6楼
邮箱:seqhealth@seqhealth.cn
电话:027-65527552
热门服务
关注康测公众号











