-
-
-
核心技术 >
检测服务 >
产品/解决方案>
-
Nanopore cDNA全长转录组
产品介绍
自1977年Sanger测序技术问世发展以来,测序技术通过解释生物遗传密码,在分子生物学研究中发挥了关键作用。全长转录组测序(full-Length transcripts sequencing)是基于牛津纳米孔技术公司(Oxford Nanopore Technologies,ONT)三代测序平台的高通量测序技术,通过纳米孔技术对cDNA分子进行测序,能够获得完整的转录本序列信息。此技术无需打断,便可直接读取从5'端到3'端polyA尾的高质量单个RNA分子全长序列,能够准确辨别二代测序无法准确识别的可变剪接(AS)、可选择性多聚腺苷酸化(APA)、融合基因等复杂的结构信息,从而克服了二代测序技术在转录本拼接短、信息不完整方面的难点。作为领先的的三代测序技术,纳米孔测序技术具有长读长、便携、成本低等优点,广泛应用于疫情防治、疾病诊断、动植物育种等各个科学领域。
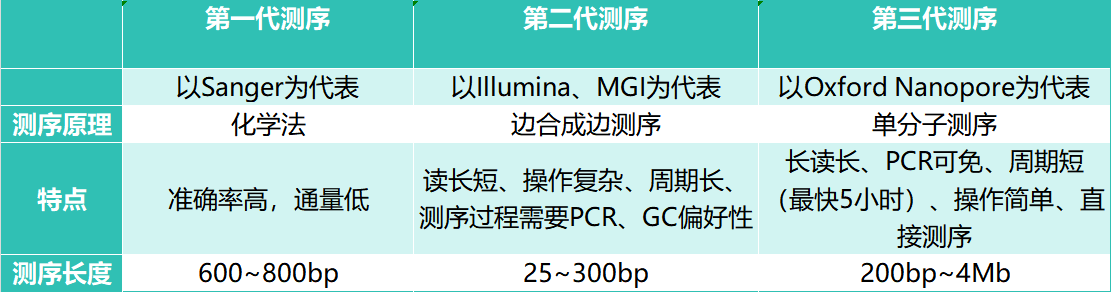
技术对比

测序原理
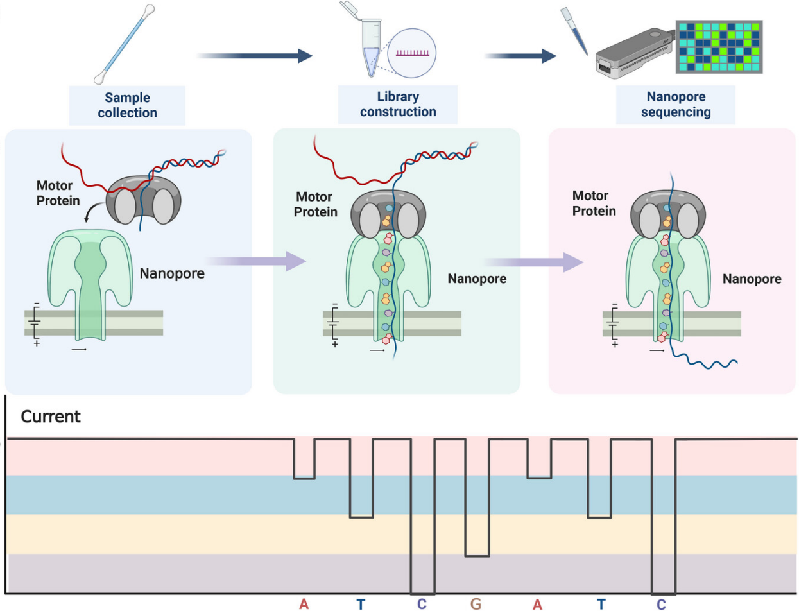
ONT测序是基于电信号识别碱基序列的三代测序技术。DNA/RNA上不同碱基化学性质存在差异,单个核酸分子在分子马达的带领下与镶嵌在生物膜上的纳米孔蛋白结合并解旋,通过纳米孔通道时,碱基造成的阻碍大小不一,因此会形成特征性离子电流变化信号。Nanopore 根据电流的大小及电流大小的变化情况,通过“递归神经网络(Recurrent Neural Network)”的复杂算法对碱基进行判读(Krishnakumar et al., 2018)。通过对这些信号进行实时检测,即可获得相应碱基类型,完成测序 。
实验流程
文库构建示意图如图所示,主要实验步骤如下: 提取total RNA; 使用Nanodrop、Qubit、琼脂糖凝胶电泳或生物片段分析仪质检; 使用引物对目标mRNA进行反转; 低循环PCR扩增全长cDNA; 加测序接头(含马达蛋白); 使用芯片进行测序。

服务流程

技术优势
- 全长读取:无读长限制(最长可达4.65Mb),一次性读取完整转录本,避免拼接错误。
- 实时测序:可实时监控测序进展,实现电子靶向测序,按需测序。
- 检测复杂事件:能够检测融合基因、可变剪接等复杂转录事件。
示例结果
我们通过Nanopore cDNA全长转录组测序技术,可以获得结果包括:
1)转录本及基因信息基本统计: 转录本和基因数目图:对转录本和基因从所有、已知和新鉴定三个方面进行统计并绘图。 转录本密度分布图:统计转录本在参考基因组上的分布情况,从外到内依次为染色体、所有转录本、已知转录本和新转录本。

2)差异表达转录本分析结果: 差异表达转录本火山图:根据各比较组的显著差异转录本, 火山图能直观展示比较组之间差异转录本的情况, 图形中, 越靠近两端的转录本, 差异程度越大。 差异表达转录本聚类热图:对差异转录本表达模式进行层级聚类, 并使用热图来呈现聚类结果。这些表达模式相近的转录本, 可能具有共同的功能或参与到共同的代谢途径及信号通路中。

3)个性化分析结果: 可变剪切分析:对转录本发生可变剪切事件情况进行统计绘制饼图(不同颜色代表不同的可变剪切事件类型),同时可进行差异分析生成差异可变剪切结果统计堆积柱形图(纵坐标表示对应类型可变剪接事件占总数的比例,横坐标分别表示差异可变剪切数量,和显著差异的可变剪接数量) 转录因子分析:使用对应数据库进行转录因子鉴定,并基于转录因子家族比例统计生成饼图。

案例分析
生物细胞中的DNA序列里面包含许多重复序列(repeated sequence),主要可分为两大类,分别是串联重复序列(也叫串接重复序列,Tandem repeat)与散置重复序列(Interspersed repeat,也被称为转座子TE,transposable element)。以往研究中,研究人员在对健康个体的人类血浆cell-free RNA测序时发现了一小部分重复序列来源的cell-free RNA (1-2%)。然而,重复序列来源的cell-free RNA对疾病的诊断潜力并不清楚。 本研究中,研究人员在二代转录组测序基础上结合三代全长测序技术,同时检测编码和非编码全长RNA(图a),从而建立了一个用于cell-free RNA定量的自定义转录组注释,该注释包含了人类基因组RepeatMasker数据库中发现的超过500万个重复序列。研究人员进一步将单个重复序列的RNA信号聚合到亚家族水平,从而将重复序列特征的数量从超过500万个减少到大约15,000个。与常规RNA-seq相比,结合三代全长测序技术显著提高了胰腺癌患者cell-free RNA数据中reads的比对率(图b)。胰腺癌患者和健康个体的cell-free RNA中比对上的重复序列的亚家族的总数之间没有显著差异(图c)。通过PCA分析(图d、e),对胰腺癌患者和健康个体的cell-free RNA的数据进行无重复序列或有重复序列分析,均能较好地实现无监督疾病识别。接下来,研究人员进行了差异表达分析(图f),发现Alu亚家族是胰腺癌患者游离RNA中富集程度最高的重复序列信号,与健康个体相比,胰腺癌患者中AluY、AluSc、AluSg7、AluSc8、AluSx3和AluSg亚家族元件富集程度最高。 研究人员进一步定量分析了肺癌、肝癌、食管癌、结直肠癌、胃癌患者和健康对照人群的cell-free RNA-seq数据,发现每种癌症类型cell-free RNA都具有特定的重复序列模式,证明了三代全长测序技术在RNA液体活检中用于癌症诊断的普遍性和适用性。

Reggiardo R E, Maroli S V, Peddu V, et al. Profiling of repetitive RNA sequences in the blood plasma of patients with cancer[J]. Nature Biomedical Engineering, 2023, 7(12): 1627-1635.
Nanopore cDNA全长转录组
联系我们
地址:武汉市江夏区东湖新技术开发区高新六路99号南山·光谷自贸港A栋6楼
邮箱:seqhealth@seqhealth.cn
电话:027-65527552
热门服务
关注康测公众号











