-
-
-
核心技术 >
检测服务 >
产品/解决方案>
-
差异基因中找不到关注的基因,如何解决?
差异基因中找不到关注的基因,如何解决?
康测云分析平台之GSEA富集分析
组学研究中,有时我们会遇到这样的情况:GO/KEGG富集分析中出现了我们比较感兴趣的通路,但是却找不到预期关注的基因。进一步检查发现关注的基因甚至不在差异基因列表里!究其原因,竟然是关注的基因在组间的表达差异不显著。在反复检查,排除掉实验和分析问题后,我们不得不作出抉择:更换靶标基因(意味着前期的分子实验可能打水漂了)?调整分析参数(得衡量可接受度,阈值放宽后也不一定出的来)?重新再做实验(时间成本高,结果可能还和这次一样)?那么,还有其它的可行策略么?有!不妨试一下GSEA富集分析。
富集分析方法对比

基因富集分析(Gene Set Enrichment Analysis,GSEA),是一种基于基因表达数据的计算方法,用于确定先验定义的一组基因是否在两种生物学状态之间显示出统计学上显著的一致性差异。这里,需要重点解释一下:首先,“先验定义的一组基因”指的是进行GSEA富集分析需要先提供一个基因集,可以使用GSEA自带的MSigDB数据库中定义的基因集或者进行自定义,基因集中包含关注的基因。其次,“两种生物学状态”可以简单理解为分组信息,比如实验组和对照组,通常和表型密切联系。最后,“一致性差异”指的是基因集中的基因在两种生物学状态中呈现出的协同变化趋势,或者说基因集整体的表达模式更接近于哪种生物学状态。总的来说,进行GSEA富集分析,我们需要提供基因表达矩阵、分组信息和定义的基因集。随后,依据与生物学状态的关联度(即排序指标,比如FC、P Value等),生成基因排序列表。接着,从上到下遍历排序列表中的基因,如果当前基因也存在于定义的基因集中,则增加富集分数。如果不存在,则减少富集分数。增加/减少富集分数的多少由排序指标决定。生成的富集分数折线图中最高峰顶或最低峰谷位置的富集分数作为基因集的富集得分,富集得分反映基因集在两种生物学状态中的协同趋势(上调/下调)。而基因集中包含的基因在排序列表中的位置分布(上端富集/底层富集)展现了基因集与哪种生物学状态更相关。
GSEA
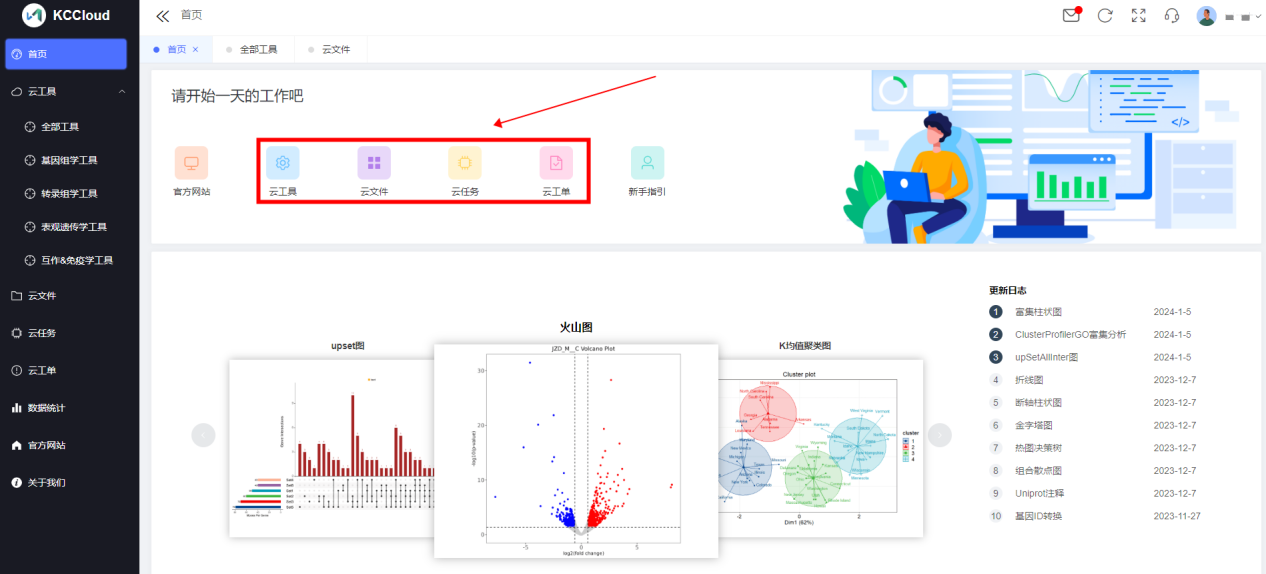
康测科技云工具已超过100种,并可实现快速查找。云文件中数据1次上传即可反复使用,并且支持多种工具同时调用。云任务中可实时追踪任务状态。云工单可随时提交问题和建议。康测云分析平台极大地提高了科研效率,让客户省心省力。
康测科技云分析平台
康测科技GSEA富集分析云工具的详细教程请参考:
工具操作十分简捷,方便客户快速入手。
最后,我们简单讲解一下GSEA富集分析的结果。
GSEA富集分析结果
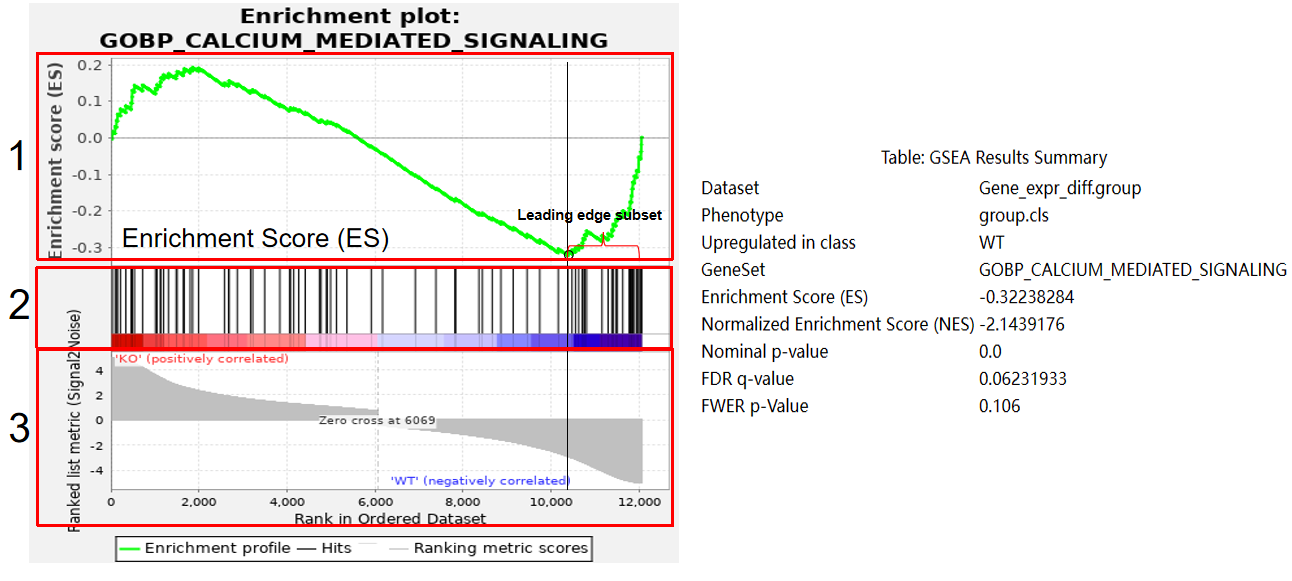
如上图所示,大体可分为三个部分。第一部分是富集分数折线图。通过从上到下遍历排序列表中的基因,计算每个基因位置的富集分数,从而生成折线图。折线图中峰顶或峰谷位置的富集分数为该基因集的富集得分(ES)。Leading edge subset指的是对ES贡献最大的基因群,当ES>0时,通常是出现在峰值得分之前的基因集合。而当ES<0时,则是出现在峰值得分之后的基因集合(简单讲就是峰顶左侧或峰谷右侧)。Leading edge subset是需要重点关注的,其中的基因在某种生物学状态下具有更显著的生物学意义。第二个部分,用黑色的线条标记了基因集中的基因出现在排序列表中的位置。也就是说,排序列表和基因集中都有的基因才会有线条标记。排序列表中从上往下的基因排列对应着第二部分从左往右的线条排列,下方红蓝相间的colorbar表示的是排序指标值的大小。红色部分的基因在左侧分组中高表达(图中是KO组),蓝色部分的基因在另一个分组中高表达(图中是WT组)。第三部分,是排序列表中基因的Rank值分布,每个基因对应的信噪比(基因排序方法,康测科技目前支持Signal2Noise、tTest、Ratio_of_Classes和log2_Ratio_of_Classes四种方法)以灰色面积展示。
关于康测
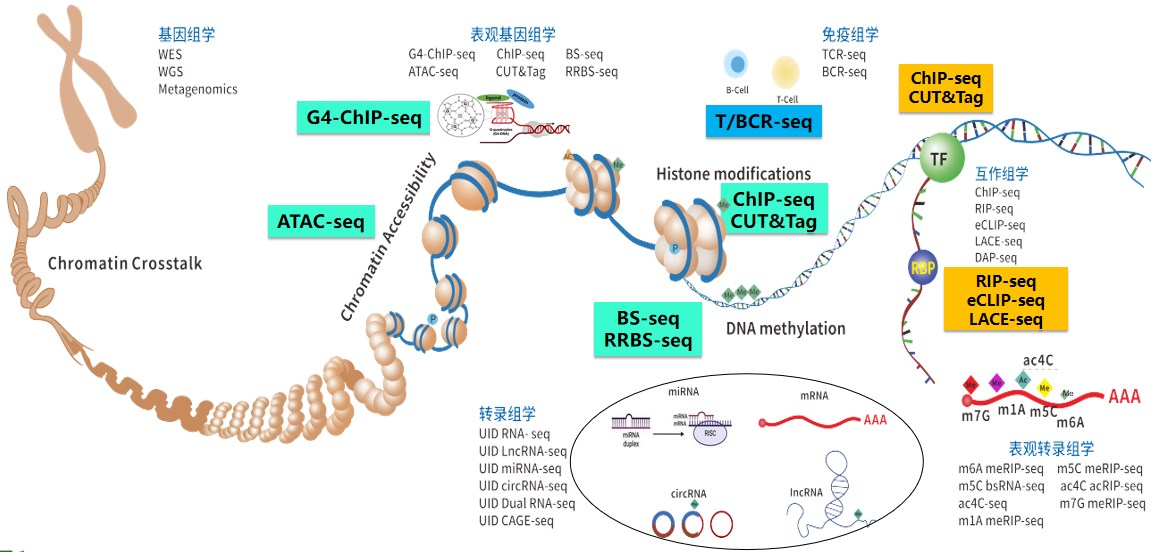
康测致力于先进组学技术的开发和在生物医学研究领域的应用,建立了涵盖基因组学、表观基因组学、转录组学、表观转录组学、免疫组学和互作组学的全面组学服务体系。而在医学检测方面,康测基于自主研发的SMP(Stranded Multiplex PCR)靶向测序技术,可提供检测灵敏度和特异性均为100%的MRD一站式自动化解决方案。
康测提供全面的基因表达调控研究工具
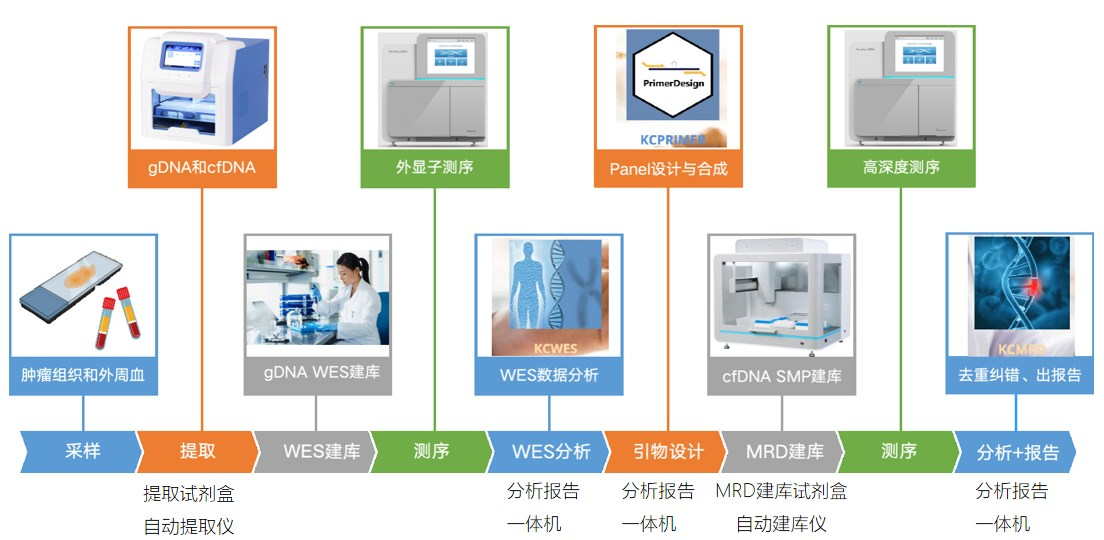
康测提供高度自动化MRD一站式解决方案
联系我们
地址:武汉市江夏区东湖新技术开发区高新六路99号南山·光谷自贸港A栋6楼
邮箱:seqhealth@seqhealth.cn
电话:027-65527552
热门服务
关注康测公众号











