Nature Plants这篇够全面,详解病菌侵染后六种主流RNA修饰变化,并诱导翻译重编程
Nature Plants:这篇够全面,详解病菌侵染后六种主流RNA修饰变化,并诱导重编程
由病原菌(如稻瘟菌)引发的植物病害会对农作物(如水稻)的生长和产量构成重大威胁。Nature Plants|这篇够全面,详解病菌侵染后六种主流RNA修饰变化,并诱导翻译重编程面对病原菌侵染,植物会迅速在转录后水平上进行重编程。其中,mRNA的选择性翻译成为影响蛋白质丰度并进而调节植物先天免疫的重要机制。此外,RNA修饰也全局调控着mRNA的翻译,并广泛影响植物生长发育和胁迫响应等众多生物学过程。然而,病原菌侵染植物后RNA修饰的动态变化以及其在植物免疫反应中的作用仍未完全了解。本期,小编为大家带来一篇最新的探究稻瘟菌侵染水稻后,水稻中六种主流RNA修饰(m1A、2'O-Nm、ac4C、m5C、m6A和m7G)变化以及ac4C主导翻译重编程从而调控水稻稻瘟病抗性的高分机制文章。


2024年9月24日,四川农业大学陈学伟、王静和陆翔老师团队在Nature Plants(IF 15.8)上发表了题为“Dynamics of epitranscriptomes uncover translational reprogramming directed by ac4C in rice during pathogen infection”的研究文章,揭示了稻瘟菌侵染水稻后,水稻中翻译组学和表观转录组学变化,并表征了m1A/m5C/m6A/m7G/ac4C/2,O-Nm 6种RNA修饰的分布模式和motif特征。研究发现感染稻瘟菌后,水稻中ac4C修饰动态变化与mRNA翻译重编程密切相关。此外,ac4C富集于密码子第三位,可能通过增强与同源tRNA的互作来提高翻译效率。感染稻瘟菌后,水稻中ac4C writer OsNAT10表达增加,并催化OsAOC、OsERF77 mRNA上ac4C修饰,从而促进茉莉酸的生物合成以及增强植物免疫反应。研究表明ac4C在水稻抵抗稻瘟菌感染中发挥了关键作用。
康测科技为本研究提供UMI m1A/m6A/m7G meRIP-seq、UMI ac4C acRIP-seq、UMI ac4C-seq和UMI m5C bsRNA-seq技术服务,全面绘制感染稻瘟菌后水稻中m1A/m6A/m5C/m7G/ac4C修饰图谱,表征了各修饰类型分布模式和motif特征。
研究思路

研究结果
一、稻瘟菌侵染引发水稻中翻译重编程
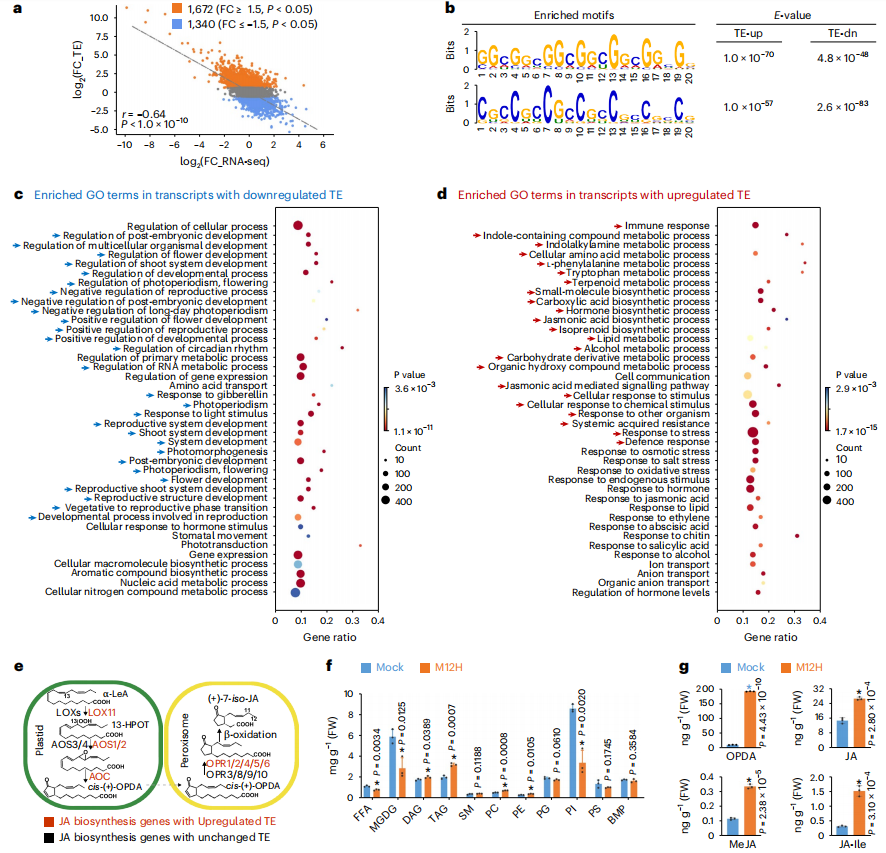
为了探究稻瘟菌侵染后水稻中转录组学和翻译组学变化,作者对感染M. oryzae分离株Zhong10-8-14(M12H)或对照(Mock)的水稻叶片进行RNA-seq和Ribo-seq。结果显示:M. oryzae侵染后,水稻中共有1,672个转录本翻译效率提高(TE-up)以及1,340个转录本翻译效率降低(TE-dn)(Fig. 1a)。并且,TE-up和TE-dn转录本均富集GGN和CNN motif(Fig. 1b)。GO分析表明,翻译效率降低的转录本在生长和发育相关的term中富集(Fig. 1c)。而翻译效率提高的转录本则在免疫反应和新陈代谢相关的term中富集,包括脂质代谢以及脂质衍生物茉莉酸(JA)生物合成过程(Fig. 1d,e)。
已知脂质代谢和JA生物合成过程与稻瘟病抗性有关。因此,作者还检测了感染M12H或Mock的水稻叶片中脂质和JA含量。作者发现M. oryzae侵染后,水稻中脂质底物,包括单半乳糖二酰甘油(MGDG)和磷脂酰肌醇(PI)的含量显著减少,但磷脂酰胆碱(PC)和磷脂酰乙醇胺(PE)的含量略有增加(Fig. 1f)。同时,12-氧代植物二烯酸(OPDA)、JA、MeJA和茉莉酸-异亮氨酸(JA-Ile)的含量显著增加(Fig. 1g)。综上所述,M. oryzae侵染引发水稻中翻译重编程并通过激活JA 生物合成来增强免疫反应。

二、稻瘟菌侵染引发的水稻中翻译变化与ac4C有关
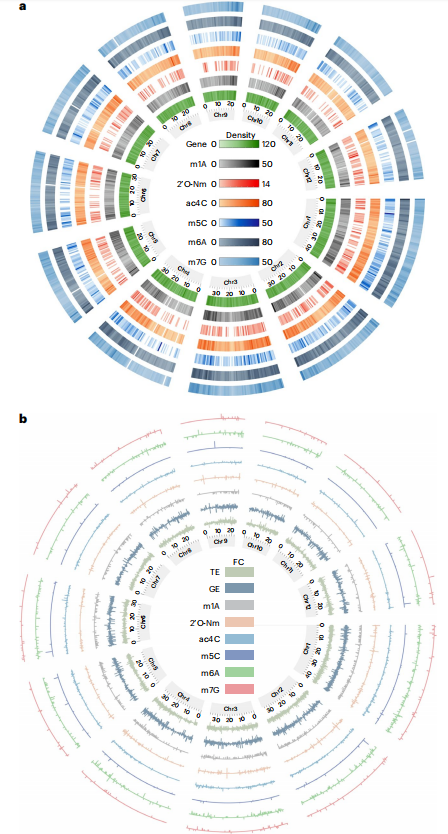
RNA修饰在调节mRNA稳定性和翻译,进而调控植物发育和抗逆方面发挥关键作用。作者利用m1A/m6A/m7G meRIP-seq、2'O-Nm-seq、ac4C-seq和m5C bsRNA-seq绘制了水稻中6种主流RNA修饰图谱(Fig. 2a)。作者发现共有8,519、1,386、4,705、1,820、10,825和9,433个转录本分别携带m1A、2'O-Nm、ac4C、m5C、m6A和m7G修饰。其中,有665、543、965、78、553和92个转录本分别显示m1A、2'O-Nm、ac4C、m5C、m6A和m7G修饰存在显著差异(Fig. 2b)。

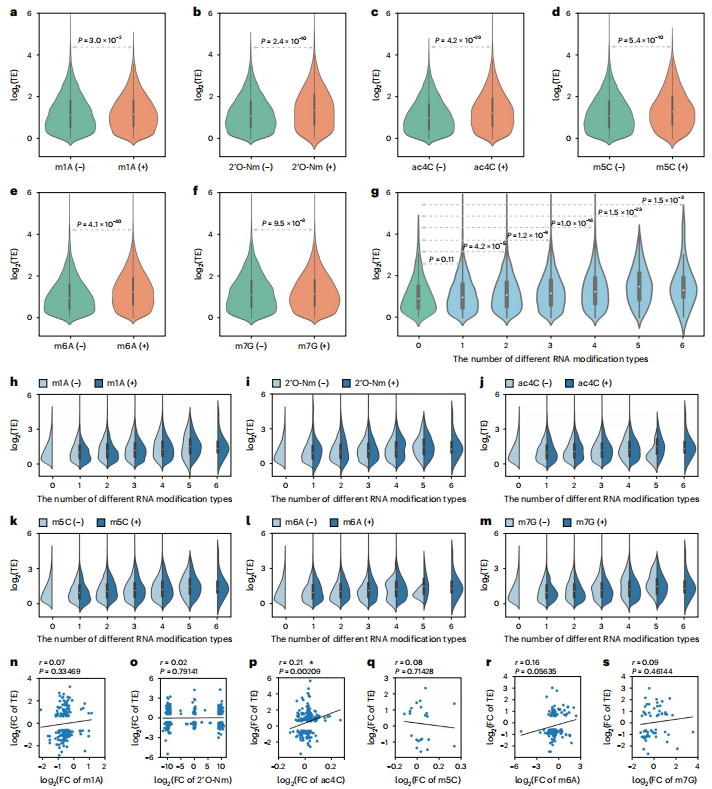
研究中的6种RNA修饰均与mRNA翻译(Fig. 3a-f)和丰度呈正相关。并且,转录本上RNA修饰类型的数量越多,翻译效率(Fig. 3g-m)和丰度就越高。然而,M. oryzae侵染后,水稻中只有ac4C修饰变化与翻译效率变化在全转录组水平上显著相关(Fig. 3p),但其他修饰类型相关性不太显著(Fig. 3n,o,q-s)。这意味着ac4C修饰主要介导M. oryzae侵染后水稻中翻译效率变化。

三、ac4C在密码子第3位富集并提高翻译效率
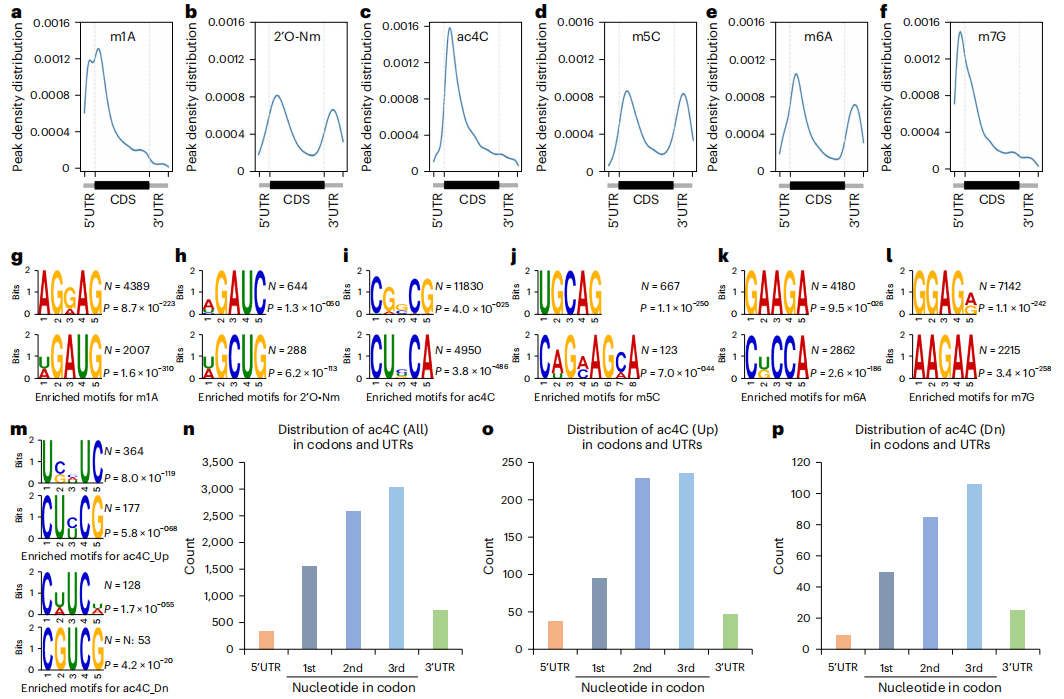
作者进一步分析了RNA修饰在转录本上5'UTR、CDS和3'UTR区的密度分布(Fig. 4a-f)。作者发现4种分布模式:1)m1A在5'UTR和CDS区富集。2)2'O-Nm、m5C和m6A在3'UTR和CDS区富集。3)ac4C在CDS区富集。4)m7G在5'UTR区富集。
接着,作者利用MEME Suite对富集motif进行分析(Fig. 4g-l)。m1A、m6A和m7G peak富集富含GA的motif,但彼此各有不同(Fig. 4g,k,l)。m1A富集GAUG motif(Fig. 4g),m6A富集CNCCA motif(Fig. 4k),2'O-Nm富集NGAUC和NGCUG motif(Fig. 4h),m5C富集富含CAG的motif(Fig. 4j),而ac4C富集CNNCN motif(Fig. 4i,m)。
此外,ac4C主要位于编码区,并在密码子第3位上显著富集(Fig. 4n-p),这一特点可能增强与摆动位置上同源tRNA的互作。总之,ac4C在密码子第3位富集,通过增强与同源tRNA的互作,从而提高翻译效率。

四、OsNAT10通过添加ac4C来增强水稻稻瘟病抗性
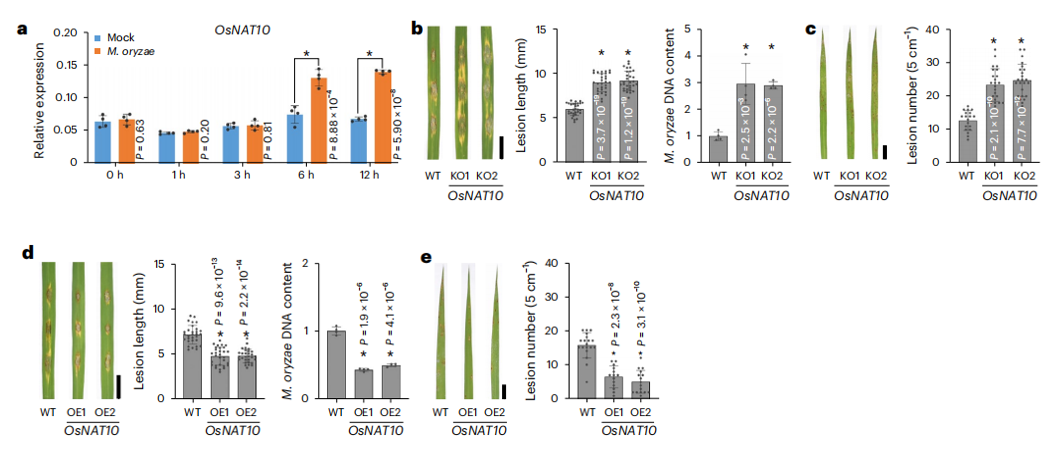
作者检测了水稻乙酰转移酶OsNAT10(LOC_Os12g07300)的转录水平,发现M. oryzae侵染6-12h后水稻中OsNAT10表达上调(Fig. 5a)。同时,mRNA上ac4C丰度显著增加(P = 0.01617),而总RNA上ac4C丰度增加不显著(P = 0.06445)。随后,作者构建了OsNAT10敲除(OsNAT10-KO)植株。与野生型(WT)植株相比,OsNAT10-KO植株中总RNA和mRNA上ac4C丰度显著降低。此外,ac4C RIP-qPCR证实OsNAT10调控M. oryzae侵染后水稻中特定转录本上ac4C修饰。
接种M. oryzae后,作者发现与WT植株相比,OsNAT10-KO1和OsNAT10-KO2植株上病斑更长、真菌生物量更高以及病斑数量更多,这意味着敲除OsNAT10导致水稻稻瘟病抗性减弱(Fig. 5b,c)。相反,OsNAT10-OE1和OsNAT10-OE2植株比WT植株的病斑更短、真菌生物量更低以及病斑数量更少(Fig. 5d,e)。上述结果表明OsNAT10通过调节mRNA上ac4C水平正向调节水稻稻瘟病抗性。

五、OsNAT10通过影响翻译来调控JA生物合成
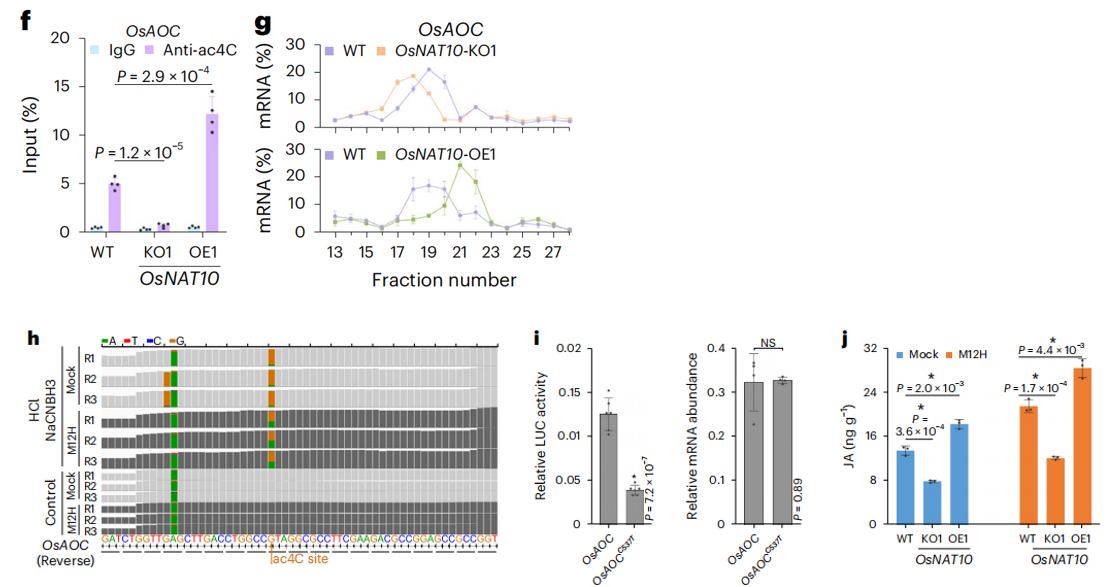
丙二烯氧化物环化酶OsAOC(LOC_Os03g32314),编码JA生物合成的限速酶,可以调节水稻幼苗发育和稻瘟病抗性。作者发现M. oryzae侵染后OsAOC的翻译效率和ac4C水平提高。并且,OsNAT10-KO1植株中OsAOC上ac4C水平降低,而OsNAT10-OE1植株中OsAOC上ac4C水平升高(Fig. 5f)。
多聚核糖体分析揭示,与WT植株相比,OsNAT10-KO1植株中OsAOC翻译效率降低,而OsNAT10-OE1植株中OsAOC翻译效率提高(Fig. 5g)。作者进一步构建突变OsAOC mRNA(OsAOCC537T),其模拟去乙酰化的OsAOC mRNA,但不改变氨基酸(Fig. 5h)。OsAOCC537T-LUC mRNA来源的荧光素酶活性低于OsAOC-LUC mRNA,但转录本丰度没有明显影响(Fig. 5i)。为了研究OsNAT10在JA生物合成中的作用,作者检测了M. oryzae侵染后WT、OsNAT10-KO1和OsNAT10-OE1植株中JA含量。与WT植株相比,OsNAT10-KO1植株中JA生物合成减少,而OsNAT10-OE1植株中JA生物合成增强(Fig. 5j)。综上所述,OsNAT10催化OsAOC mRNA上ac4C修饰,进而提高 OsAOC mRNA的翻译效率,从而促进JA生物合成过程。

除了OsAOC,作者还发现OsERF77(LOC_Os04g52090)(编码ERF/AP2转录因子,负调节水稻生长),也在稻瘟菌侵染后表现出ac4C水平和翻译效率提高。与WT植株相比,OsNAT10-KO1植株中OsERF77 mRNA的ac4C水平和翻译效率降低,而OsNAT10-OE1植株中OsERF77 mRNA的ac4C水平和翻译效率提高(Fig. 6a,b)。同样的,作者构建了突变OsERF77 mRNA(OsERF77C318T),其模拟去乙酰化的OsERF77 mRNA,而不改变氨基酸(Fig. 6c)。
OsERF77C318T-LUC mRNA来源的荧光素酶活性低于OsERF77-LUC mRNA,而对于转录本丰度,OsERF77C318T-LUC与OsERF77-LUC处于相似水平(Fig. 6d)。为了探究OsERF77在稻瘟病抗性调节中的作用,作者构建了OsERF77敲除(OsERF77-KO)和OsERF77过表达(OsERF77-OE)植株。与WT植株相比,OsERF77-KO植株表现出稻瘟病抗性减弱,而OsERF77-OE植株表现出稻瘟病抗性增强(Fig. 6e-h)。
接着,作者利用DAP-seq鉴定到9555个OsERF77结合靶基因(Fig. 6i)。对WT和OsERF77-KO植株进行RNA-seq,获得336个差异表达基因(DEG)。再将DAP-seq和RNA-seq数据取交集,得到52个重叠基因。GO分析表明这52个基因在核糖体生物发生、rRNA加工、翻译和细胞对刺激的反应等过程富集(Fig. 6j)。作者进一步检测可能参与细胞对刺激反应的OsERF77靶基因,包括OsAOS2(LOC_Os03g12500)、OsLOX3(LOC_Os03g49350)、OsGLIP1(LOC_Os06g03890)和OsALDH2b(LOC_Os06g15990)在OsERF77-KO和OsERF77-OE植株中的表达水平。结果显示OsERF77负调控OsGLIP1和OsALDH2b表达,但正调控OsAOS2和OsLOX3表达(Fig. 6k,l)。
此外,作者成功验证了OsERF77与OsAOS2的结合(Fig. 6m-o),OsERF77也直接结合OsGLIP1和OsALDH2b。而OsAOS2是JA生物合成中的重要基因。并且,与WT植株相比,OsERF77-KO植株中JA含量降低,而OsERF77-OE植株中JA含量提高(Fig. 6p)。综上所述,OsNAT10提高OsERF77 mRNA的ac4C水平和翻译效率,OsERF77结合OsAOS2并增强OsAOS2的表达,最终促进JA生物合成。
参考文献
Xiang Lu, Yao He, Jinqiao Guo, et al. Dynamics of epitranscriptomes uncover translational reprogramming directed by ac4C in rice during pathogen infection[J]. Nature Plants, 2024











